シングルセル空間トランスクリプトーム解析

シングルセル空間トランスクリプトーム解析とは
1細胞ごとに組織内の位置情報を保持したまま遺伝子発現解析が可能です。組織切片上の転写産物(RNA)を1分子ごとに検出し、細胞境界情報と合わせて解析することで、個々の遺伝子が発現する位置(組織内の細胞)と量を計測することができます。そのため、シングルセルRNA-seqのような1細胞遺伝子発現解析法では失われていた空間的な位置情報を保ったまま、シングルセルトランスクリプトーム解析を実施することが可能です。
病変部を構成する細胞の種類や発現情報の取得だけでなく、病変部周辺にエンリッチした細胞に有意に発現する遺伝子の探索、目的細胞に隣接する細胞の種類や特徴的に発現する遺伝子の同定が可能です。また、相互作用する細胞ペアとその分子(リガンドと受容体)、シグナル伝達パスウェイを推定することもできます。細胞間相互作用解析などの網羅的な遺伝子発現情報が求められる解析においては、シングルセルRNA-seqデータ等との統合解析が有用です。
空間オミクス解析の1種である空間トランスクリプトーム解析法は、3つのタイプに分けられます(詳細はこちら)。手法により得られる結果の空間解像度や検出遺伝子数は様々であるため、研究目的に合わせて最適な手法を選択することが重要です。
用途
- 組織内に局在する各細胞タイプの推定(未知の細胞タイプ検出を含む)
- 組織内に局在する各細胞タイプで特徴的に発現する遺伝子の同定
- 組織内における細胞間相互作用(細胞ペア、リガンド及び受容体、パスウェイ)の推定
- 組織内の位置情報を考慮したpseudotime解析による細胞系譜(分化経路)推定
- 組織内の位置情報を考慮したRNA velocty解析による細胞動態の評価
- 目的細胞の近傍にエンリッチする細胞タイプや発現遺伝子の同定
- 目的細胞における各遺伝子の細胞内局在の検出
※関連する解析サービス
スポット型空間トランスクリプトーム解析 >
シングルセルRNA-seq解析 >
ホルマリン固定後のシングルセルトランスクリプトーム解析 >
高感度なシングルセル完全長total RNA-seq解析 >
シングルセルレパトア解析+シングルセルRNA-seq解析 >
このような方に
- シングルセルRNA-seq解析では細胞や発現遺伝子の位置情報が得られずお悩みの方
- 参考論文と同じような解析を自身のサンプルで実施したい方
- 自身の研究目的に最適な実験デザインの検討から始めたい方
- 簡易ソフトでは希望の解析ができず、目的の結果の取得にお困りの方
- 空間トランスクリプトームとscRNA-seqデータの統合解析をご希望の方
- 論文や公共データベースで公開されているデータを用いた解析をご希望の方
当社の特徴
- 共同研究タイプの受託解析サービス(定型的な受託解析ではありません)
- 経験豊富な専門技術者がお問い合わせから直接対応
- 各種計測法や各種計測データの解析に対応
10x Genomics社 Xenium In Situシステム*1
NanoString社 CosMx SMIシステム*2
Vizgen社 MERSCOPEシステム*3等 - 生物学的意義を踏まえたデータ解析サービス(詳細はこちら)
- 解析結果の統計学的および生物学的解釈のサポート
※「当社が選ばれる5つの理由」はこちら
空間トランスクリプトーム解析法の種類
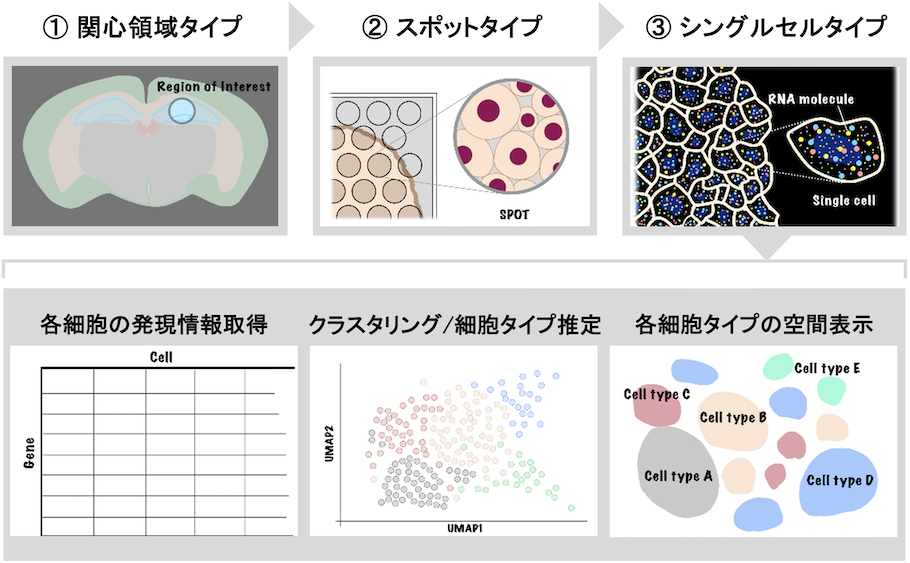
空間オミックス解析の1種である空間トランスクリプトーム解析法は、3つのタイプに大別されます。手法ごとに得られる結果が大きく異なるため、研究目的に合わせて最適な手法を選択することが重要となります。
①関心領域(ROI)タイプ
レーザーやUVなどで選択した組織切片上の関心領域について、通常のRNA-seq解析を実施する手法です。病変部や腫瘍部などの関心領域における、バルク(集団の平均値)の網羅的な発現情報を取得可能です。しかしながら、個々の細胞の発現情報や空間的な位置情報を得ることはできません。
②スポットタイプ >
基盤上に配置したスポットごとに、網羅的な遺伝子発現解析を実施する手法です。空間的な位置情報を保持しながら遺伝子発現解析が可能です。空間分解能の違いにより複数のタイプに分けられます。ひとつはVisium*1で、スポットのサイズやスポット間の距離により、1スポットに数十個程度の細胞が含まれたり、解析されない細胞が一定数生じるタイプです。もうひとつはVisium HD*1で、細胞とほぼ同サイズのスポットで、スポット間の隙間のない高解像度タイプです。これらのタイプは研究目的や予算により適切な使い分けが必要です。
③シングルセルタイプ >
組織切片上のRNAを1分子ごとに検出し細胞境界情報と併せて解析することで、1細胞ごとにトランスクリプトーム解析を実施する手法です。個々の遺伝子が発現する位置(組織内の細胞)と量を計測する遺伝子発現解析ができます。通常の(空間情報を含まない)シングルセル解析の結果を基に注目する遺伝子を選択し、それらの遺伝子(細胞)の空間情報を取得することを主な目的に使用します。網羅的な遺伝子発現解析も可能ですが、検出遺伝子数については数百~数千遺伝子と網羅性に関してはまだ課題もあります。
解析例
解析例1 小腸の組織常在型メモリーT細胞分化の局所的制御
マウスの小腸急性感染モデルで取得したシングルセル空間トランスクリプトームデータ(Xenium*1データ)を使用し、LCMV感染前後における前駆細胞様TRM細胞の細胞数変化、TRM細胞の分化と遊走に関連するケモカインの局所的発現変化を検出しました。TRM(Tissue-Resident Memory T, 組織常在メモリーT)細胞:リンパ系以外の組織に常在するCD8+T細胞。
小腸絨毛に存在する腸管上皮細胞に特徴的な遺伝子の発現分布
小腸絨毛に存在する腸管上皮細胞に特徴的な遺伝子の発現分布

小腸内腔には絨毛(villi)と、絨毛間のくぼみに位置する陰窩(crypt)と呼ばれる構造が存在します。小腸絨毛に存在する細胞タイプに特徴的に発現する遺伝子の空間分布を、遺伝子毎に各色の点で示しました。
小腸絨毛に存在する腸管上皮細胞に特徴的な遺伝子の発現分布(細胞毎の発現量)
小腸絨毛に存在する腸管上皮細胞に特徴的な遺伝子の発現分布(細胞毎の発現量)
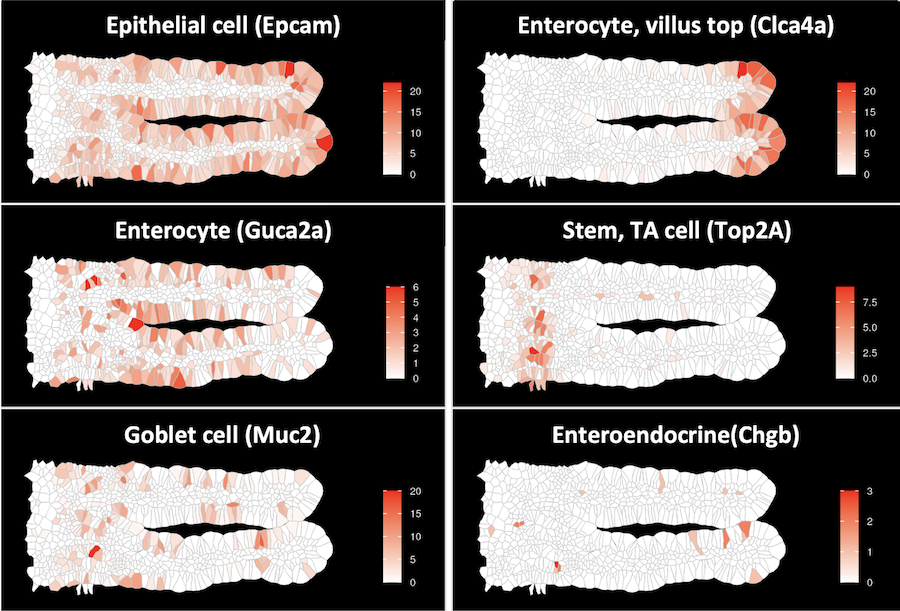
前図で示した各遺伝子の発現分布とそれに紐づく発現量を基に、個々の遺伝子の細胞毎の発現量をヒートマップで表示しました。各パネルの上部に細胞タイプ(遺伝子名)を示しました。絨毛先端部には分化した吸収上皮に特徴的なClca4aを発現する細胞(上段)が分布し、陰窩周辺部には増殖中の細胞に特徴的なTop2aを発現する幹細胞およびTransit amplifying(TA)細胞(中段)が分布していました。絨毛には吸収上皮の他に、Muc2を発現する杯細胞(下段)や、Chgbを発現する少数の腸管内分泌細胞(下段)の分布が見られました。
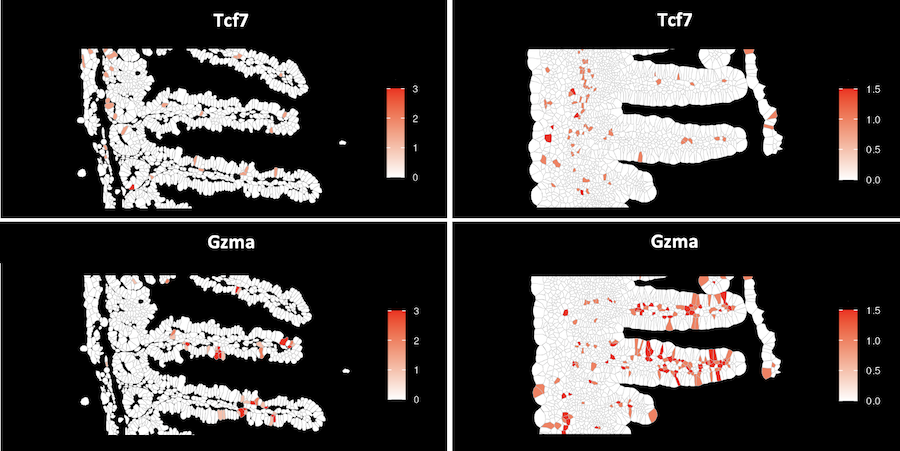
小腸絨毛に存在する免疫細胞に特徴的な遺伝子の発現分布(細胞毎の発現量)
小腸絨毛に存在する免疫細胞に特徴的な遺伝子の発現分布(細胞毎の発現量)
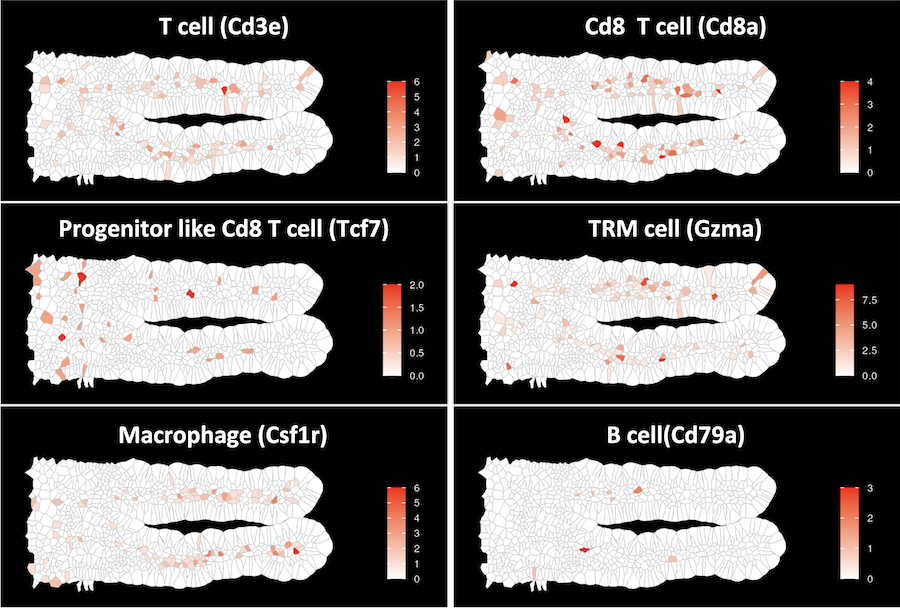
免疫細胞に特徴的な遺伝子の細胞毎の発現量をヒートマップで示しました。前駆細胞様TRM細胞に特徴的なTcf7を発現するT細胞(中段)は絨毛基底部に分布し、分化したTRM細胞に特徴的なGzmaを発現するT細胞(中段)は絨毛先端部側に分布していました。絨毛内部全体にはマクロファージや少数のB細胞(下段)が分布していました。
急性感染による「TRM細胞の分化・遊走に関連するCxcl10」の発現変化
急性感染による「TRM細胞の分化・遊走に関連するCxcl10」の発現変化
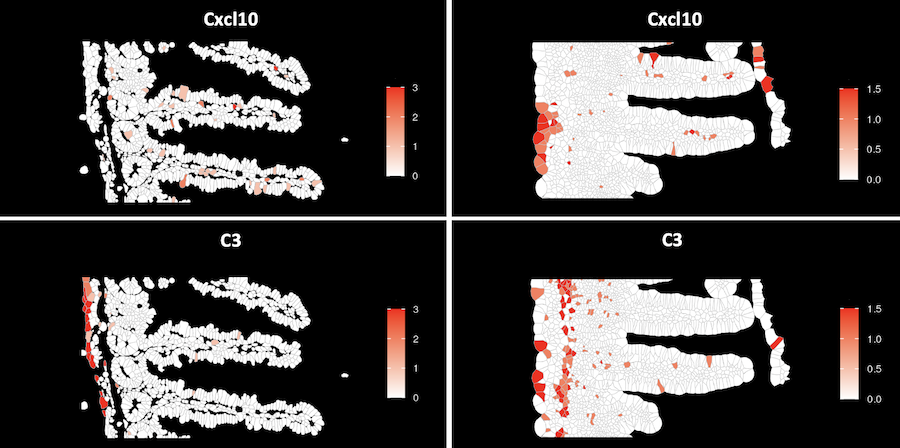
小腸急性感染モデルを用いて、TRM細胞の分化と遊走に関連するCxcl10の発現分布を、感染前(左列)および感染6日後(右列)で比較しました。Cxcl10(上段)は感染により筋層で発現増加が見られ、一方、一部の線維芽細胞で発現が見られるC3(下段)は、感染の有無に関わらず筋層で発現していました。感染により、筋層に分布するC3陽性線維芽細胞でのCxcl10の発現増加が示唆されました。
急性感染によるCxcl10発現部位における前駆細胞様TRM細胞の細胞数変化
急性感染によるCxcl10発現部位における前駆細胞様TRM細胞の細胞数変化
感染前(左列)および感染6日後(右列)の小腸絨毛における、前駆細胞様TRM細胞(上段)および分化したTRM細胞(下段)の分布を示しました。感染6日後には前駆細胞様TRM細胞の絨毛基底部での増加が見られました。筋層でのCxcl10の発現と絨毛基底部での前駆細胞様TRM細胞の増加は一過性で、感染後の時間経過により消失しました。一方、Cxcl10の受容体であるCxcr3をノックアウトしたマウスでは、前駆細胞様TRM細胞の増加が抑制されました。
シングルセル空間トランスクリプトーム解析はこのように、多様な細胞や遺伝子発現が関与する生命現象に対して、多種多様な遺伝子の空間情報を同時に解析する事を可能にしました。通常のシングルセル解析などの、空間情報を含まない網羅的な遺伝子発現解析結果に基づいて空間情報を取得し解釈することで、生命現象のより高度な理解が得られることが期待できます。
解析例2 ヒト肺腺がん組織における制御性T細胞(Treg)の局在
ヒト肺腺がん組織より取得したシングルセル空間トランスクリプトームデータ(Xeniumデータ*4、1細胞ごとの発現情報と位置情報)を用いて、組織内の各細胞タイプの局在推定、各細胞タイプに特徴的に発現する遺伝子の検出、およびその遺伝子(RNA分子)の細胞内局在を可視化しました。
細胞のクラスタリング結果 (497,763cells, UMAP)
細胞のクラスタリング結果 (497,763cells, UMAP)
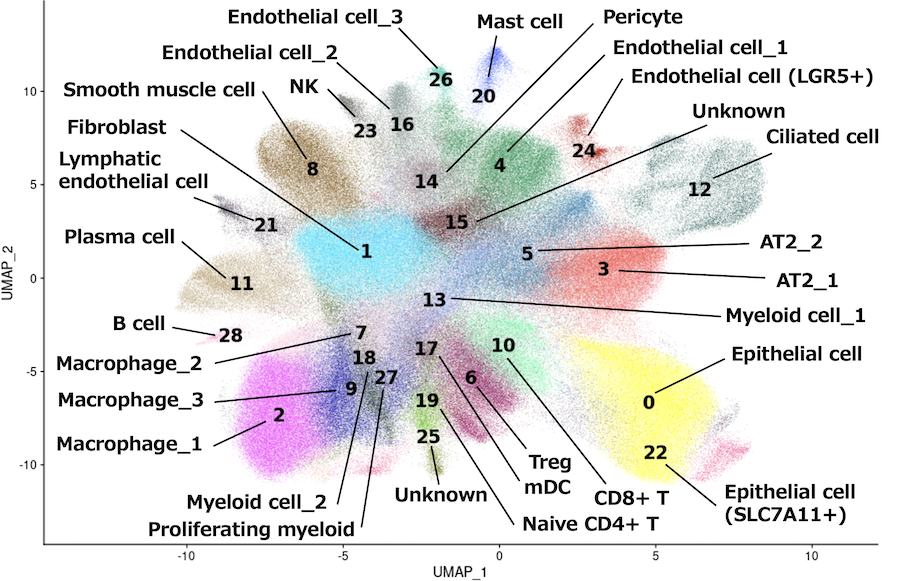
各細胞の遺伝子発現情報(発現プロファイル)を、情報のロスを極力抑えつつ低次元に変換しました。その後、発現プロファイルの類似度に基づいて細胞をクラスタリングし、その結果をUMAP*5により二次元上にプロットしました。1つの点が1個の細胞を示します。各点には細胞の発現プロファイルが紐づいており、発現が類似しているほど各点は基本的には近くに配置されます。
各細胞タイプに特徴的な遺伝子発現(Dot plot)
各細胞タイプに特徴的な遺伝子発現(Dot plot)
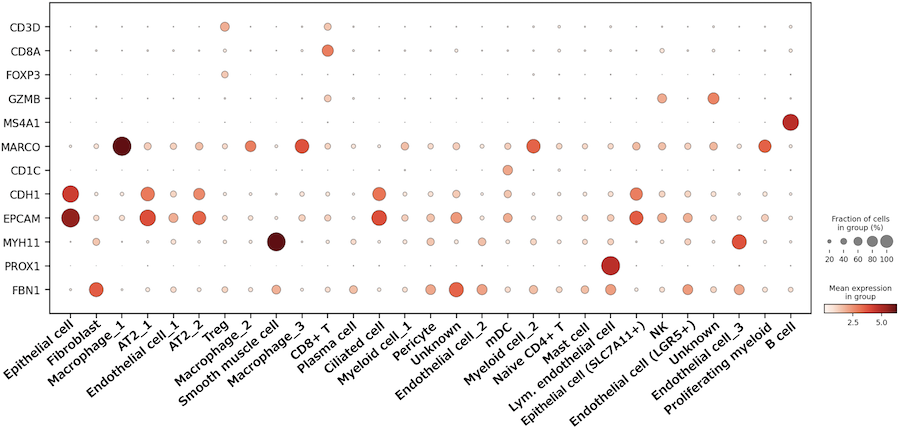
各細胞タイプ(クラスタ)に特徴的な遺伝子の発現量と発現細胞割合をドットプロットで図示しました。縦軸は遺伝子、横軸は各細胞タイプに対応しています。発現量をドットの色の濃淡で、発現している細胞の割合をドットの大きさで示しました。
各細胞の組織切片像へのマッピング結果
各細胞の組織切片画像へのマッピング結果

クラスタリング結果に基づいて細胞を色分けし組織切片画像(空間座標)にプロットすることで、組織中の各細胞タイプの局在を可視化しました。1つの点が1個の細胞を示します。
各細胞の組織切片像へのマッピング結果(細胞タイプ毎)
各細胞の組織切片画像へのマッピング結果(細胞タイプ毎)
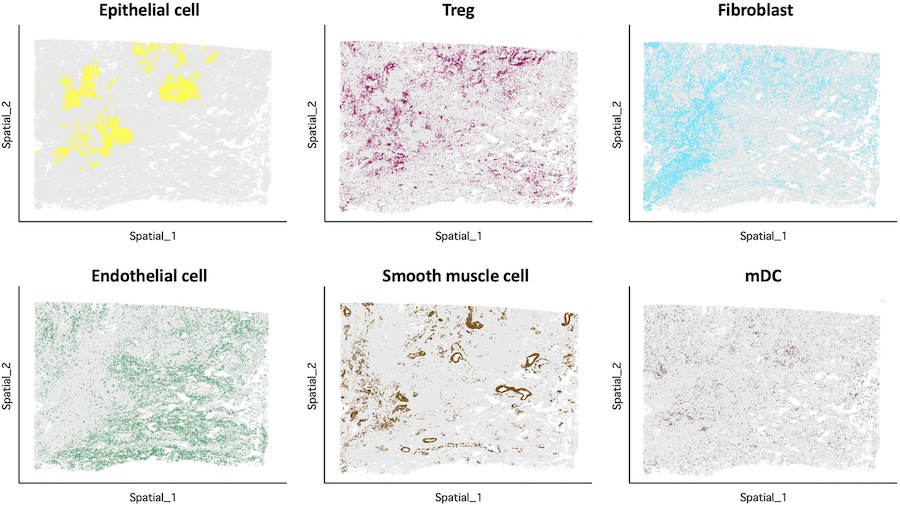
組織中の各細胞タイプの局在を細胞タイプごとに表示しました。
肺腺がん微小環境における腫瘍細胞および制御性T細胞(Treg)の局在
肺腺がん微小環境における腫瘍細胞および制御性T細胞(Treg)の局在
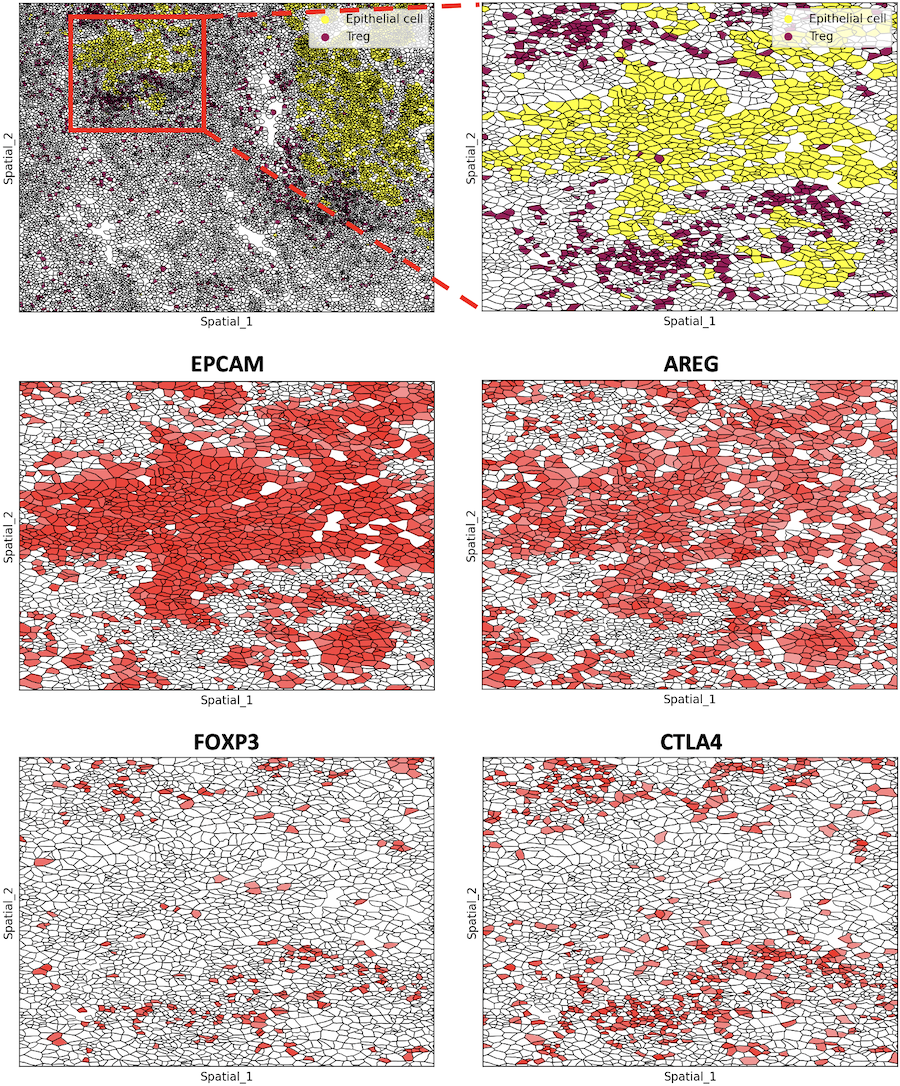
各細胞の位置情報と細胞境界線情報を基に、肺腺がん組織における上皮細胞(腫瘍細胞)とTregの分布を図示しました。また、上皮細胞、Tregの各マーカーであるEPCAM(中段左図)、FOXP3とCTLA4(下段)、さらに、TregのFOXP3の発現を誘導し免疫抑制作用を促進するAREG(中段右図)の発現量をヒートマップで示しました。発現量が高い細胞は赤色、低い細胞は白色で表示しました。 AREGは主に腫瘍細胞に発現が見られ、Tregはその細胞の周囲に局在していました。
AREGおよびFOXP3 RNA分子の空間上での発現分布
AREGおよびFOXP3 RNA分子の空間上での発現分布
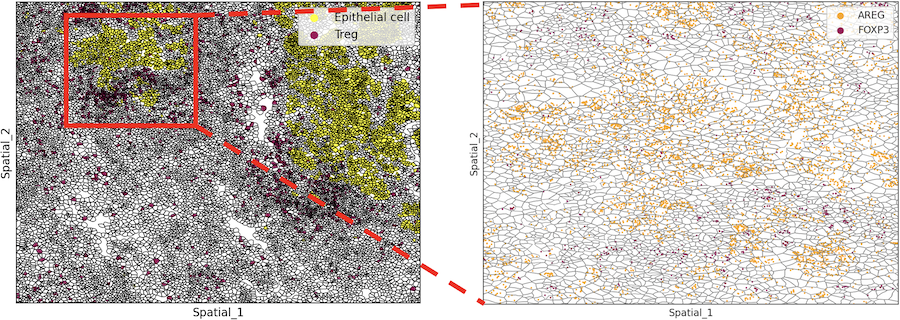
AREGおよびFOXP3の細胞内の分布をRNA1分子レベルでプロットしました(右図)。このようにシングルセル空間トランスクリプトーム解析法では、組織内の細胞の種類と分布を明らかにでき、さらに各細胞タイプに特徴的に発現する遺伝子やその遺伝子(RNA分子)の細胞内局在も検出することが可能です。
解析例3 マウス胎子肝臓の造血幹・前駆細胞に隣接して存在する細胞タイプの同定
マウス胎子肝臓を用いたシングルセル空間トランスクリプトームデータにおける検出遺伝子の不足を、同一組織で実施したシングルセルRNA-seqデータで補完し、造血幹・前駆細胞(以下、HSPC)の微小環境を構成する細胞(種類と位置)と発現する遺伝子(種類、量、位置)を高感度に検出しました。
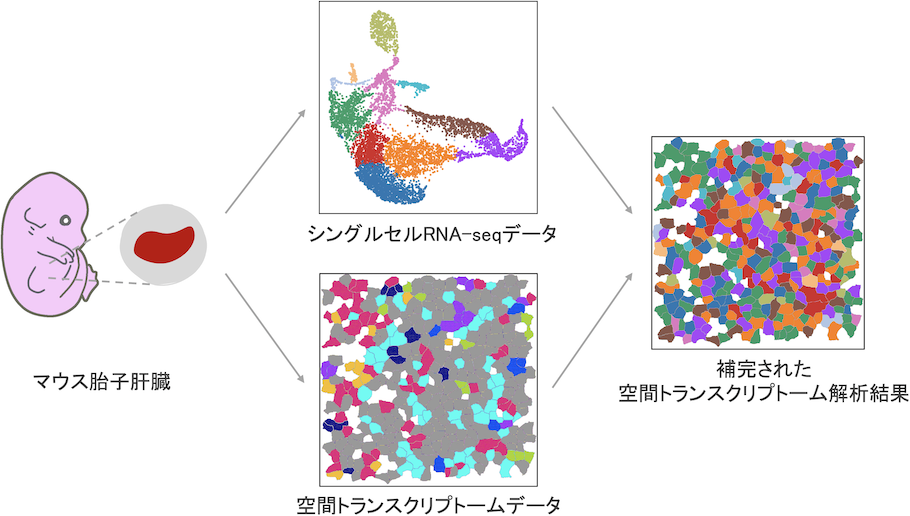
解析の概略図
解析の概略図
補完したデータによる細胞のクラスタリング結果 (40,864 cells, UMAP)
補完したデータによる細胞のクラスタリング結果 (40,864 cells, UMAP)
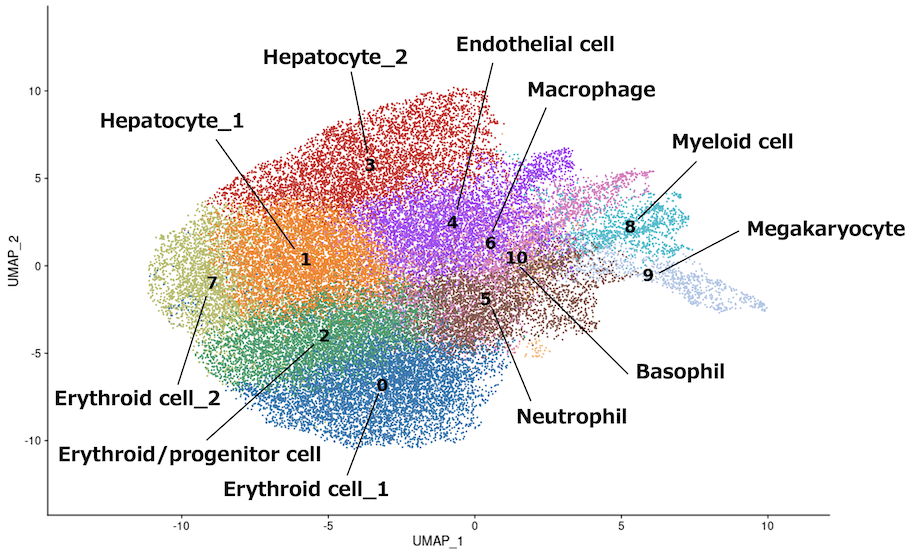
シングルセルRNA-seqデータで補完したシングルセル空間トランスクリプトームデータを用いて、遺伝子発現プロファイルの類似度に基づいて細胞をクラスタリングし、その結果をUMAPにより二次元上にプロットしました。1つの点が1個の細胞を示します。
各細胞タイプに特徴的な遺伝子の発現分布
各細胞タイプに特徴的な遺伝子の発現分布
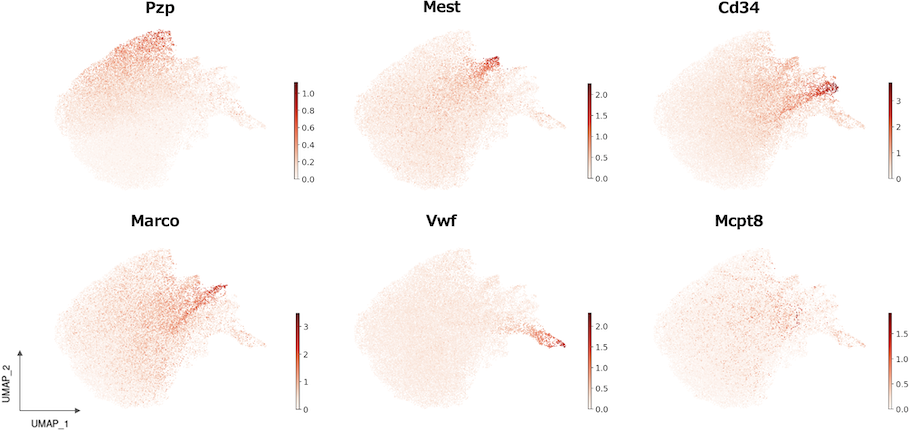
各細胞タイプで特徴的に発現している遺伝子の発現分布を図示しました。このような発現プロファイルやその他の解析をもとに、各クラスタの細胞タイプを推定しました。
各細胞の組織切片像へのマッピング結果
各細胞の組織切片像へのマッピング結果
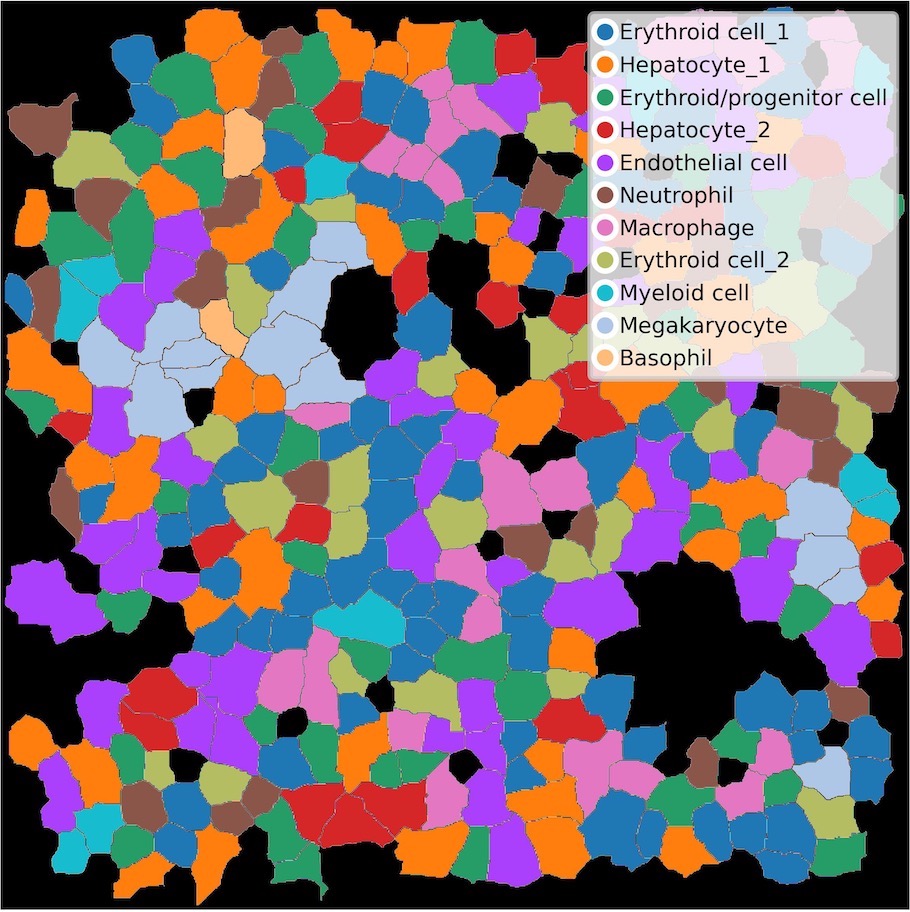
各細胞の位置情報と細胞境界線情報を基に、組織における各細胞タイプの局在を図示しました。上図は組織切片上の一部の領域を示します。
各遺伝子の組織切片における発現分布
各遺伝子の組織切片における発現分布
各細胞タイプに特徴的な遺伝子の発現量を、ヒートマップで表示しました。発現量が高い細胞は赤色、低い細胞は白色で示しました。
血管内皮細胞クラスタの再クラスタリング結果(4,900 cells, UMAP)
血管内皮細胞クラスタの再クラスタリング結果(4,900 cells, UMAP)
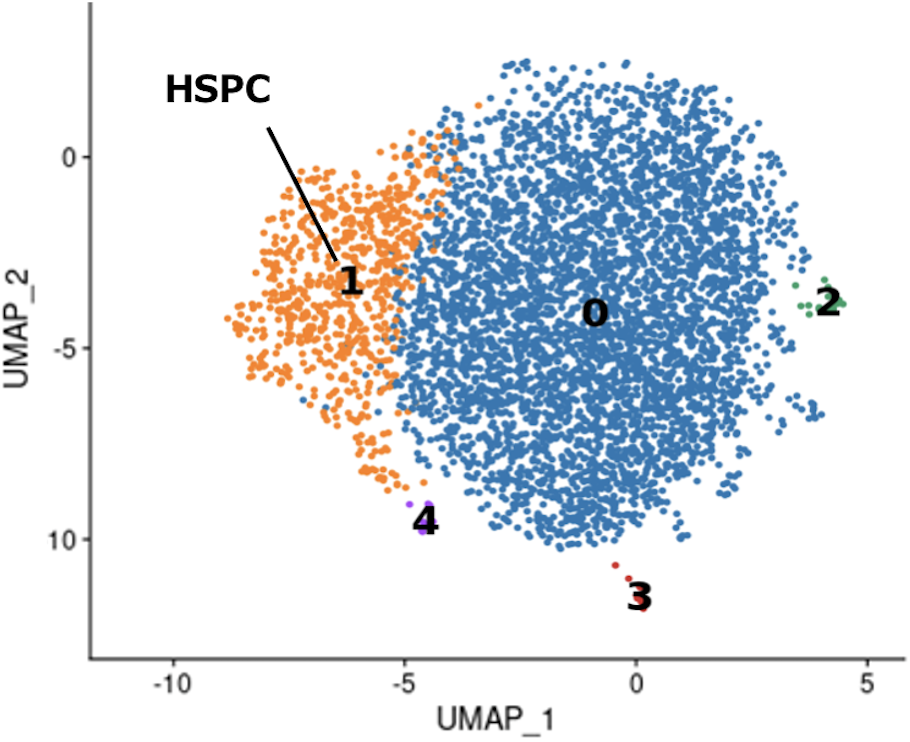
全細胞のクラスタリング結果から、血管内皮細胞と推定された細胞集団を抽出した後、再度クラスタリングを行いました。
HSPCマーカー遺伝子の発現分布
HSPCマーカー遺伝子の発現分布
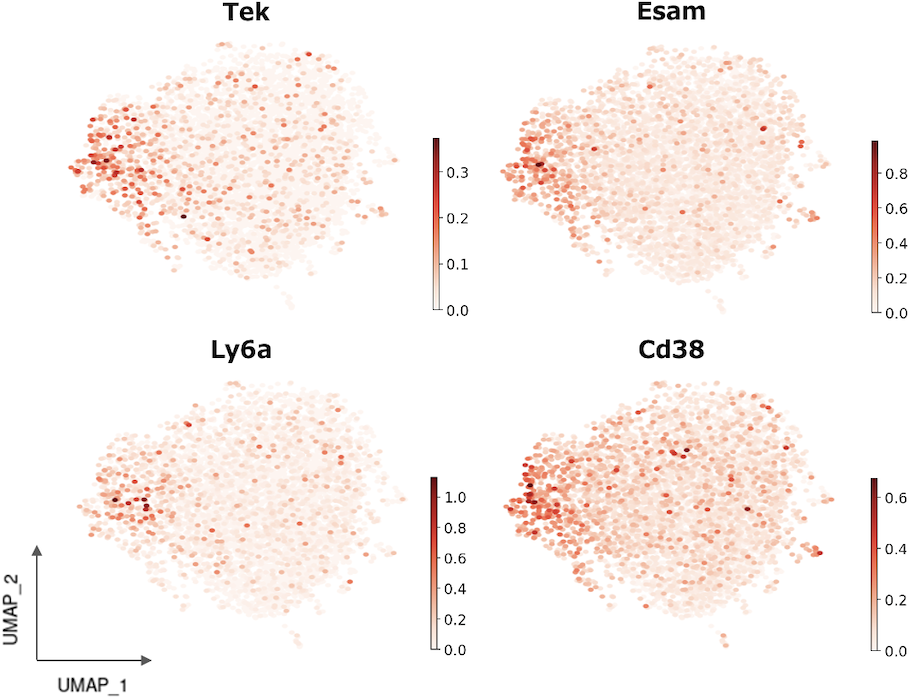
造血幹・前駆細胞(HSPC)で特徴的に発現している遺伝子の発現分布を図示しました。このようにシングルセルRNA-seqデータで補完することで、検出遺伝子数が限られるシングルセル空間トランスクリプトーム解析でも、より高感度に希少な細胞集団を検出することが可能です。
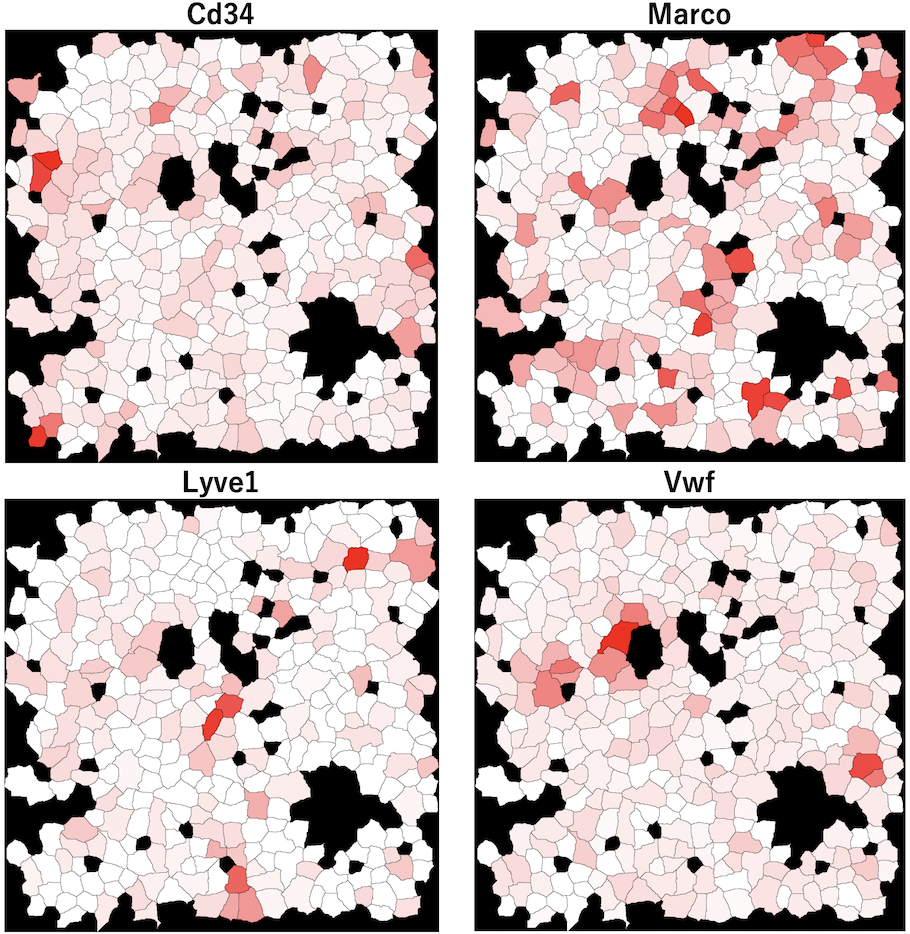
HSPC近傍における血管内皮細胞のエンリッチメント
HSPC近傍における血管内皮細胞のエンリッチメント
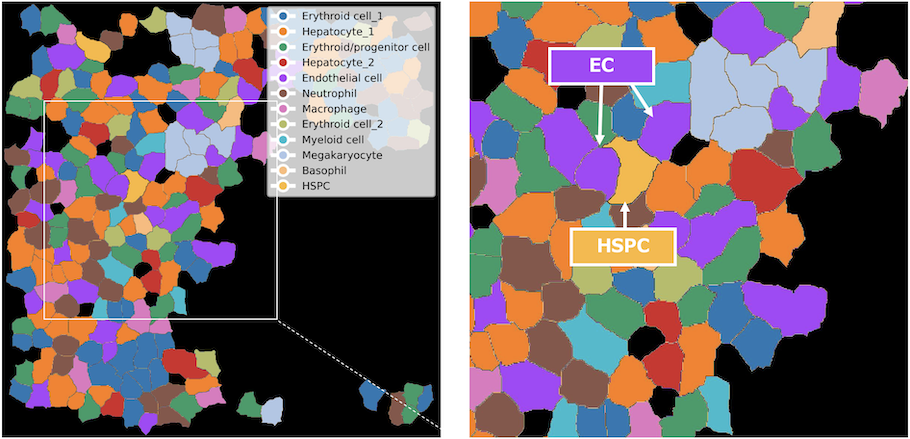
再クラスタリングにより検出したHSPCを、組織切片画像にマッピングし、HSPC近傍に存在する細胞タイプを可視化しました。左図は組織切片上の特定の領域について表示し、白枠で囲った領域を右図で拡大表示しています。多くのHSPCは、血管内皮細胞と隣接して存在していることが観察されました。EC:血管内皮細胞、HSPC:造血幹・前駆細胞
解析例4 マウス嗅球構成細胞の同定と嗅覚受容体RNA分子および糸球体の組織内局在
マウス嗅球組織切片の1細胞空間トランスクリプトームデータ(1細胞ごとの発現情報と位置情報)を用いて、組織内の各細胞タイプを推定し、遺伝子の発現分布を1細胞およびRNA1分子ごとに空間上に可視化しました。
細胞のクラスタリング結果 (27,894 cells, UMAP)
細胞のクラスタリング結果 (27,894 cells, UMAP)
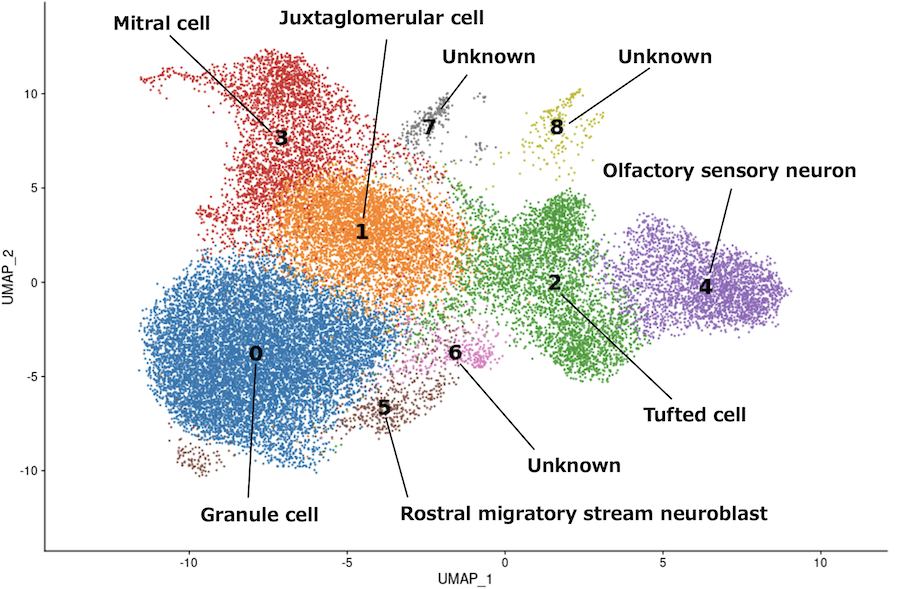
各細胞の発現プロファイルを基に細胞をクラスタリングし、その結果をUMAPにより二次元上にプロットしました。発現情報に基づいて細胞を分類することで、組織内にどのような特徴をもった細胞が存在するかを明らかにできます。
各細胞の組織切片像へのマッピング結果
各細胞の組織切片像へのマッピング結果
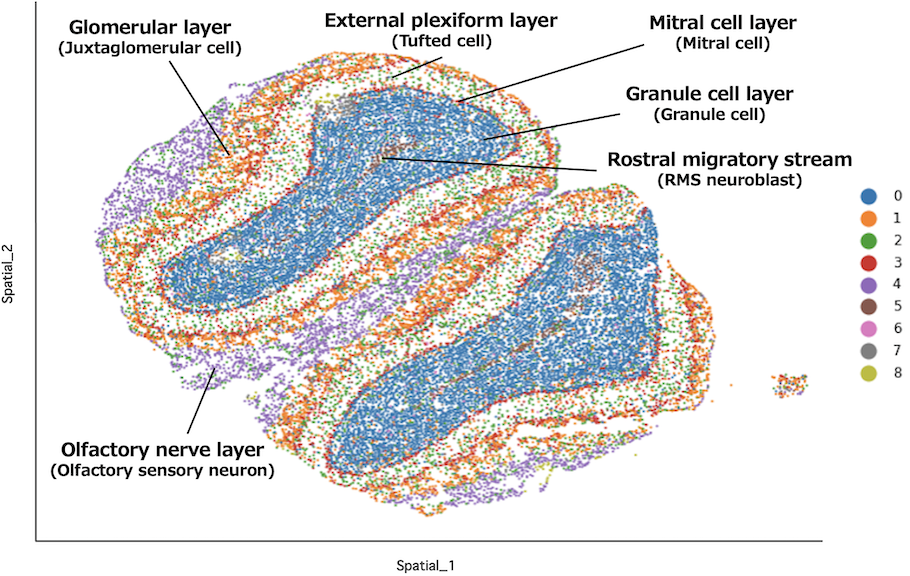
クラスタリング結果に基づいて細胞を色分けし組織切片画像(空間座標)にプロットすることで、組織中の各細胞タイプの局在を可視化しました。
各遺伝子の発現分布
各遺伝子の発現分布
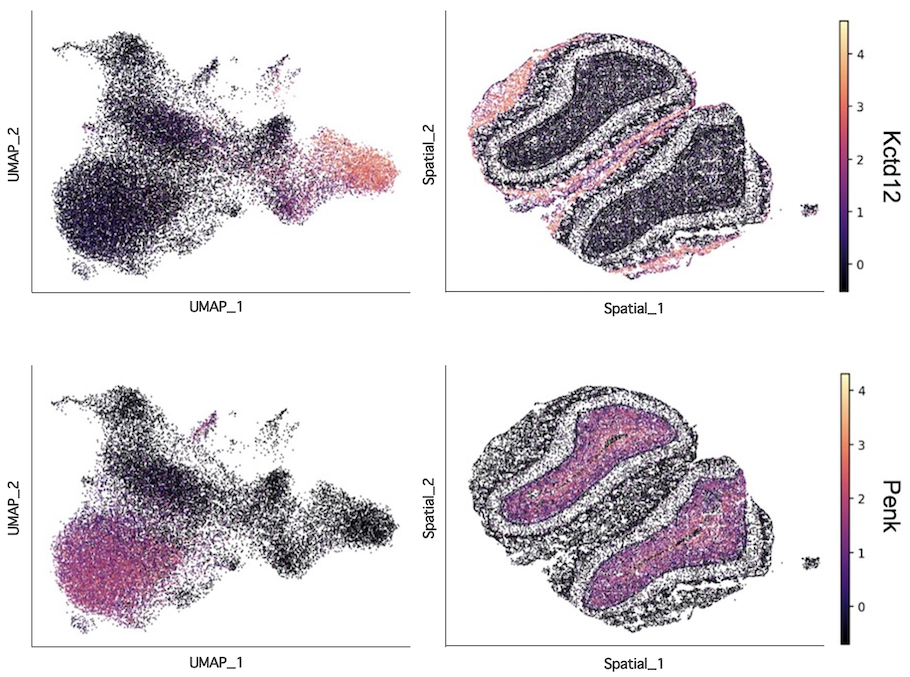
特徴的な遺伝子の発現分布を、クラスタリング結果上(左図)または組織切片上(右図)に図示しました。左図では、着目する遺伝子が特徴的に発現するクラスタ(細胞タイプ)を視覚的に確認できます。一方、右図では、着目する遺伝子が発現している細胞の組織内局在を確認することが可能です。
各RNA分子の組織切片上での発現分布
各RNA分子の組織切片上での発現分布
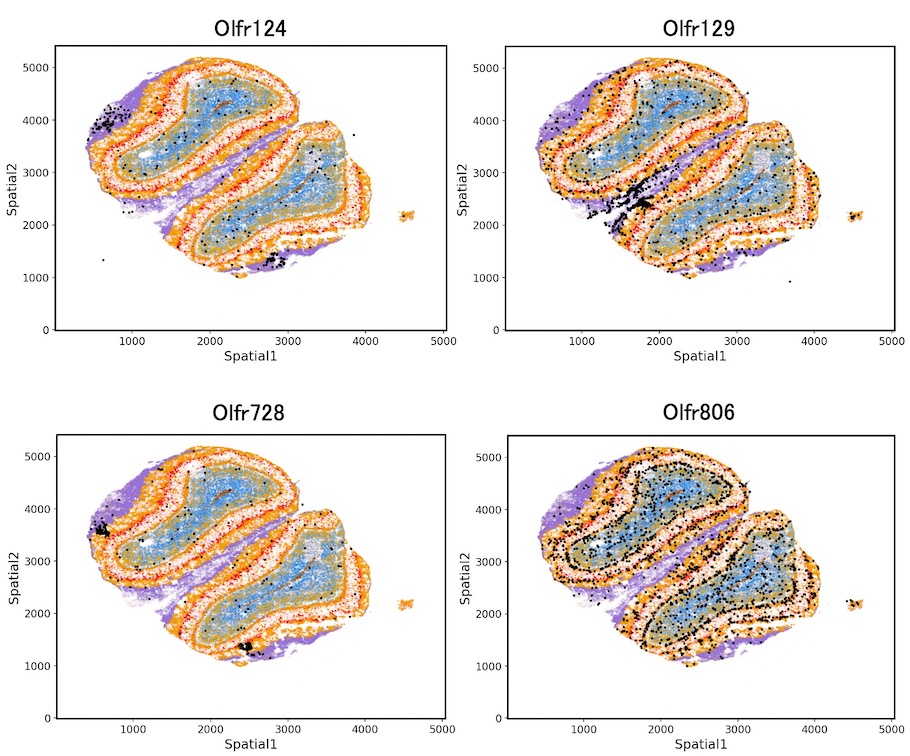
各遺伝子の発現量と発現位置を、RNA1分子レベルで組織切片画像(空間座標)にプロットしました。マウス嗅球における各細胞層のマーカー遺伝子をRNA1分子レベルで可視化し、さらに嗅覚受容体(Olfactory receptor)のRNA分子を黒点で表示しました。マウス嗅覚受容体遺伝子には約1,000種類ほどがありますが、同じ種類の嗅覚ニューロン(Olfactory sensory neuron)には、その内の1種類の嗅覚受容体が特異的に発現します。また、同じ種類の嗅覚ニューロンは、同一の糸球体に集束します。そのため、組織内において1分子単位で嗅覚受容体RNAの発現位置を明らかにし、同じ嗅覚受容体分子が局在する領域を特定することにより、糸球体の存在する位置が推定できます。
解析フロー
イメージング 各種計測データ 発現量推定
クラスタリング
細胞タイプ推定
組織切片へのマッピング
高次解析 解析結果
受入物
| ① 組織切片 / ブロックから解析 | ② 各種計測データから解析 | |
|---|---|---|
| 受入物 |
|
|
| 生物種 | ヒトまたはマウス | ― |
| 受入条件 |
【 FFPE切片の場合 】
【 FFPEブロックの場合 】
|
【 計測法 】
※上記のほか、様々な計測データに対応
|
| その他 |
【 FFPEブロックの場合 】
|
以下の情報をお知らせください
|

解析条件
- 遺伝子パネル : ヒト100~6,000遺伝子、マウス247~5,000遺伝子
- データ解析 : 研究目的に沿った最適な手法で実施します
※研究目的や対象組織により、最適な計測法や遺伝子パネル(遺伝子数)が異なります。
納品物
- 解析報告書
- 細胞のクラスタリング画像データ
- 各細胞タイプの空間分布画像データ
- 各遺伝子の1細胞単位の発現分布画像データ(クラスタリング結果上の分布、空間分布)
- 各遺伝子のRNA1分子単位の発現分布画像データ(空間分布)
※実際の納品物はご依頼内容により変わります。
価格・納期
ご依頼の解析内容により異なりますので、詳細はこちらよりお問い合わせください。
ご注意事項
受入物を品質検査した結果、解析をお引き受けできない場合があります。
お問い合わせ
こちらよりお問い合わせ下さい。
専門技術者が原則24時間以内にご連絡します。(土日祝日は除く)
参考文献
Reina-Campos, M. et al. "Tissue-resident memory CD8 T cell diversity is spatiotemporally imprinted" Nature 639, 483–492(2025). doi: 10.1038/s41586-024-08466-x
I-Hao Wang. et al. "Spatial transcriptomic reconstruction of the mouse olfactory glomerular map suggests principles of odor processing" Nat Neurosci 25, 484(2022). doi: 10.1038/s41593-022-01030-8
*1 10x Genomics社の商品名または登録商標です。
*2 NanoString Technologies社の商品名または登録商標です。
*3 Vizgen社の商品名または登録商標です。
*4 10X Genomics社より計測データをご提供頂きました。
*5 Uniform Manifold Approximation and Projection(次元削減法の1種)
