シングルセルRNA-seq解析
Single cell RNA-seq(scRNA-seq)解析とは
1細胞ごとに転写産物(RNA)の種類と量を網羅的に検出します。各細胞の遺伝子発現プロファイルを元に細胞集団を分類し、亜集団に特徴的な発現情報を取得できるため、細胞集団に含まれる細胞タイプ、割合、そして各細胞タイプに特徴的に発現する遺伝子を同定することが可能です。そのため、組織を構成する様々な細胞の機能や状態(細胞分化、増殖、老化など)を詳細に明らかにすることができます。
細胞集団の平均的な遺伝子発現量を計測するBulk RNA-seq解析とは異なり、大多数の細胞の発現情報に埋もれて検出が困難な希少細胞における発現変化も検出可能です。また、発生過程や細胞リプログラミング過程における細胞系譜(cell lineage)を詳細に追跡し、系譜に沿って発現が変化する遺伝子を同定できるのも特徴のひとつです。さらに、がんを始めとした様々な微小環境において、相互作用する細胞タイプ、リガンドと受容体、シグナル伝達パスウェイの推定にも活用できます。シングルセル解析の代表的な手法であるシングルセルRNA-seq解析は、その他にも遺伝子制御ネットワーク(GRN)や転写因子活性の予測など様々な研究用途に使用されます。
※受託解析サービスの詳細はこちら
用途
- 細胞集団に含まれる細胞タイプの推定*1(未知の細胞タイプ検出を含む)
- 細胞集団に含まれる各細胞タイプで特徴的に発現する遺伝子の同定
- 群間における各細胞タイプでの発現比較(発現変動遺伝子の検出)
- 多数の細胞中に混在する少数細胞の発現変化の検出
- Pseudotime(擬時間、擬似時間)解析による細胞分化経路の推定
- RNA velocity解析による細胞動態の推定
- 細胞間相互作用(細胞種ペア、リガンド-受容体、パスウェイ)の推定
- 各細胞タイプにおける転写因子活性の予測
※関連する解析サービス
ホルマリン固定後のシングルセルトランスクリプトーム解析 >
高感度なシングルセル完全長total RNA-seq解析 >
シングルセルレパトア解析+シングルセルRNA-seq解析 >
シングルセル空間トランスクリプトーム解析 >
スポット型空間トランスクリプトーム解析 >
このような方に
- 参考論文と同じような解析を自身のサンプルで実施したい方
- 自身の研究目的に最適な実験デザインの検討から始めたい方
- 簡易的な解析による結果と生物学的な仮説との間に相違があり、結果の解釈にお困りの方
- 共同研究先の対応スピードや一方向的な情報科学的解析手法にお悩みの方
- 最適なデータ解析手法や適切な統計手法の選択と実施に不安がある方
- 論文や公共データベースで公開されているデータを用いた解析をご希望の方
当社の特徴
- 共同研究タイプの受託解析サービス(定型的や簡易的な受託解析ではありません)
- 研究目的を踏まえ論文化を志向した高品質な解析サービス
- ウェット実験とデータ解析の両分野の経験豊富な専門技術者が直接対応
- 各種シングルセル化法・ライブラリ調製法や各種シークエンスデータの解析に対応
(10x Genomics社 Chromiumシステム*2、BD社 Rhapsodyシステム*3等) - 生物学的意義を踏まえたデータ解析サービス(詳細はこちら)
- 解析結果の統計学的および生物学的解釈のサポート
※「当社が選ばれる5つの理由」はこちら
解析例
- 解析例1 末梢血単核細胞のクラスタリングと特徴的に発現する遺伝子の抽出 >
- 解析例2 多発性硬化症患者脳脊髄液に特徴的な単核細胞と遺伝子発現変化の検出 >
- 解析例3 遺伝子ノックアウトにより異常を生じる少数細胞の発現変動遺伝子抽出 >
- 解析例4 がん組織浸潤T細胞に特徴的なサブタイプの同定 >
- 解析例5 視覚野神経細胞において視覚刺激により発現変化した遺伝子の網羅的検出 >
- 解析例6 抗原提示細胞の分化経路推定(trajectory analysis) >
- 解析例7 RNA velocity解析による細胞動態の推定 >
- 解析例8 エナメル芽細胞分化に伴って発現が変化する遺伝子群の抽出 >
- 解析例9 誘導法の違いによる人工多能性幹細胞(iPS細胞)誘導経路の検証 >
- 解析例10 疲弊したCAR-T細胞で活性化する転写因子の推定 >
- 解析例11 心臓組織の細胞老化による細胞間相互作用の変化の検出 >
解析例1 末梢血単核細胞のクラスタリングと特徴的に発現する遺伝子の抽出
ヒト末梢血単核細胞(PBMC)のシングルセルRNA-seqデータより、細胞亜集団の検出、各亜集団で特徴的に発現する遺伝子の抽出を行いました。
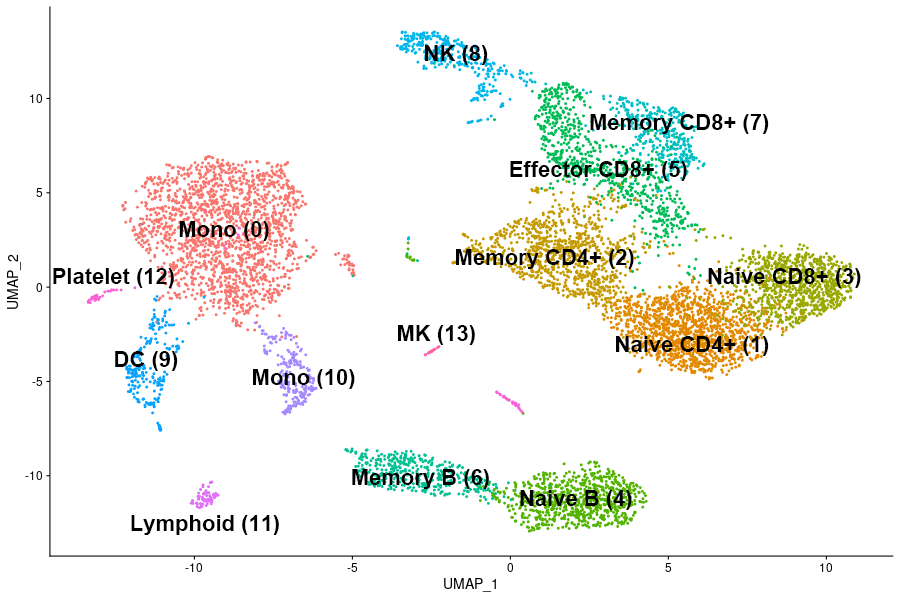
細胞のクラスタリング結果 (8,381 cells, 1群、n=1, UMAP)
細胞のクラスタリング結果 (8,381 cells, 1群、n=1, UMAP)
1. 1,354 cells
2. 972 cells
3. 954 cells
4. 809 cells
5. 761 cells
6. 421 cells
7. 368 cells
8. 340 cells
9. 221 cells
10. 218 cells
11. 67 cells
12. 62 cells
13. 20 cells
高次元のデータである各細胞の網羅的な発現情報(発現プロファイル)を、PCA(主成分分析)により情報のロスを極力抑えつつ低次元に変換しました。発現プロファイルの類似度に基づいて細胞をクラスタリングし、その結果をUMAP*4により二次元上にプロットしました。1つの点が1個の細胞を示します。各点には細胞の発現プロファイルが紐づいており、発現が類似しているほど各点は基本的には近くに配置されます。UMAP1とUMAP2(x軸とy軸)の目盛は相対的な位置を示しますが、シングルセル解析においては、個々の目盛よりも結果全体を俯瞰して理解することが重要です。

| 0. | 1,814 cells | 1. | 1,354 cells | 2. | 972 cells |
| 3. | 954 cells | 4. | 809 cells | 5. | 761 cells |
| 6. | 421 cells | 7. | 368 cells | 8. | 340 cells |
| 9. | 221 cells | 10. | 218 cells | 11. | 67 cells |
| 12. | 62 cells | 13. | 20 cells |
高次元のデータである各細胞の網羅的な発現情報(発現プロファイル)を、PCA(主成分分析)により情報のロスを極力抑えつつ低次元に変換しました。発現プロファイルの類似度に基づいて細胞をクラスタリングし、その結果をUMAP*4により二次元上にプロットしました。1つの点が1個の細胞を示します。各点には細胞の発現プロファイルが紐づいており、発現が類似しているほど各点は基本的には近くに配置されます。UMAP1とUMAP2(x軸とy軸)の目盛は相対的な位置を示しますが、シングルセル解析においては、個々の目盛よりも結果全体を俯瞰して理解することが重要です。
各クラスタの発現変動遺伝子(Heatmap)
各クラスタの発現変動遺伝子(Heatmap)
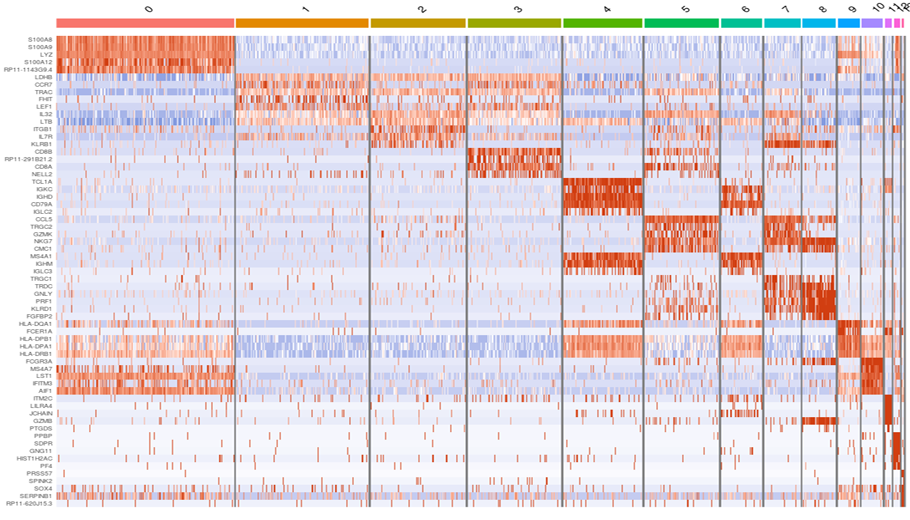

該当クラスタとその他残りのクラスタの2群間で発現比較解析を行い、統計的に有意に発現変化する遺伝子を発現変動遺伝子(DEG: Differentially expressed genes)として抽出しました。各クラスタで特徴的に発現するそれらの遺伝子をヒートマップを用いて可視化しました。縦軸は遺伝子に、横軸は各クラスタに対応しています。各クラスタ内には、そのクラスタに属する細胞1つひとつが表されています。発現量が多い遺伝子は赤色、少ないものは青色で示しました。
各クラスタの発現変動遺伝子抽出結果の一例
各クラスタの発現変動遺伝子抽出結果の一例
| cluster | gene symbol |
avg.exp cluster |
avg.exp remaining |
log FC | p value | adjusted p value |
positive in cluster |
positive in remaining |
|---|---|---|---|---|---|---|---|---|
| 0 | CD14 | 6.037 | 0.178 | 1.788 | 0 | 0 | 0.830 | 0.066 |
| 1 | CCR7 | 2.976 | 0.772 | 0.808 | 8.58E-234 | 1.57E-229 | 0.651 | 0.224 |
| 2 | IL7R | 7.074 | 2.168 | 0.936 | 8.86E-208 | 1.62E-203 | 0.829 | 0.384 |
| 3 | CD8A | 3.308 | 0.546 | 1.025 | 0 | 0 | 0.747 | 0.124 |
| 4 | CD79A | 17.334 | 1.004 | 2.214 | 0 | 0 | 0.989 | 0.146 |
| 5 | CCL5 | 38.231 | 3.586 | 2.147 | 0 | 0 | 0.954 | 0.278 |
| 6 | IGKC | 75.036 | 8.003 | 2.134 | 1.21E-199 | 2.23E-195 | 0.798 | 0.352 |
| 7 | GZMA | 10.220 | 1.919 | 1.347 | 1.04E-224 | 1.90E-220 | 0.970 | 0.207 |
| 8 | GNLY | 80.282 | 1.961 | 3.312 | 0 | 0 | 0.959 | 0.198 |
| 9 | HLA-DQA1 | 27.911 | 2.616 | 2.079 | 7.01E-276 | 1.29E-271 | 1.000 | 0.386 |
| 10 | FCGR3A | 15.407 | 0.488 | 2.400 | 3.33E-282 | 6.11E-278 | 0.986 | 0.090 |
| 11 | IRF7 | 16.354 | 0.350 | 2.554 | 5.36E-238 | 9.83E-234 | 1.000 | 0.138 |
| 12 | PPBP | 50.743 | 0.049 | 3.898 | 2.89E-119 | 5.29E-115 | 0.919 | 0.018 |
| 13 | ITGA2B | 0.532 | 0.029 | 0.398 | 9.89E-10 | 1.81E-05 | 0.300 | 0.006 |
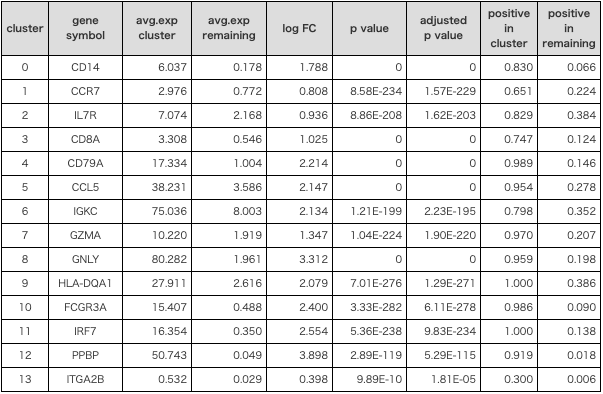

| cluster | : | クラスタ番号 |
| gene symbol | : | 遺伝子名略号 |
| avg.exp cluster | : | 該当クラスタのすべての細胞における各遺伝子の平均発現量 |
| avg.exp remaining | : | 該当クラスタ以外のすべての細胞における各遺伝子の平均発現量 |
| log FC | : | 発現変動比 |
| p value | : | 有意確率 |
| adjusted p value | : | 補正した有意確率 |
| positive in cluster | : | 該当クラスタ内で発現している細胞の割合 |
| positive in remaining | : | 該当クラスタ以外のすべての細胞の内、発現している細胞の割合 |
各クラスタに特徴的に発現する遺伝子の一部を列挙しました。このようにシングルセルRNA-seq解析では、細胞集団の組成に関する事前情報を用いることなく、クラスタリングし特徴的な遺伝子の発現変化を抽出する事ができます。
各遺伝子の発現分布
各遺伝子の発現分布
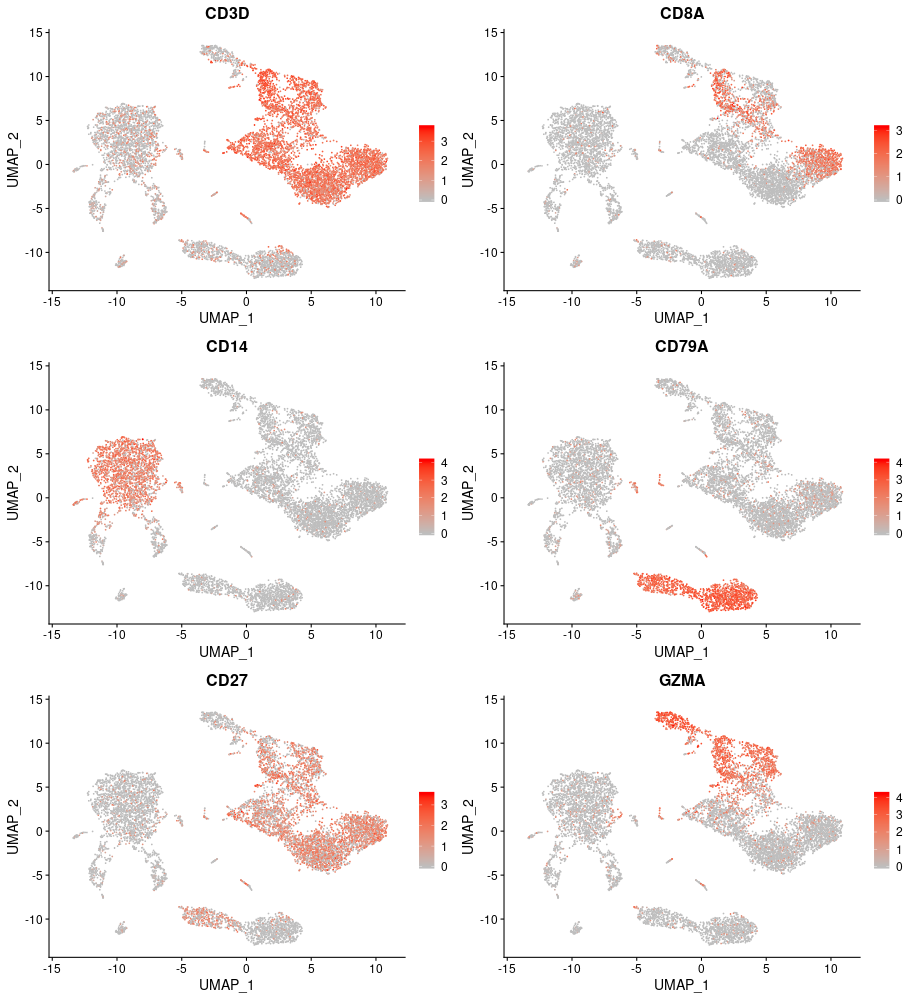
特徴的な遺伝子の発現分布を図示しました。クラスタに特徴的な遺伝子、複数のクラスタで高発現している遺伝子を視覚的に確認できます。このような発現プロファイルやその他の解析結果をもとに、各クラスタの細胞タイプを推定しました。
各クラスタにおける遺伝子の発現レベル(Violin plot)
各クラスタにおける遺伝子の発現レベル(Violin plot)
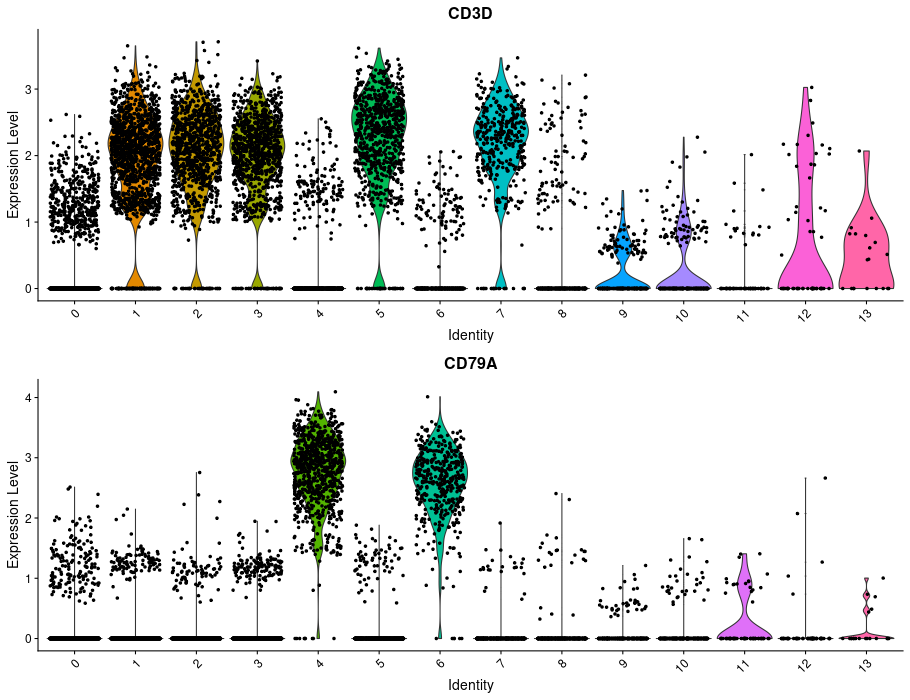

特徴的な遺伝子の発現量をバイオリンプロットで図示しました。縦軸は遺伝子発現量、横軸は細胞クラスタ、バイオリン型の幅は密度分布(プロットされた細胞の密度)を示しています。図中にプロットされた各点は個々の細胞を表しており、各クラスタの細胞ごとの遺伝子発現量を一覧できます。
解析例2 多発性硬化症患者脳脊髄液に特徴的な単核細胞と遺伝子発現変化の検出
脳脊髄液中の単核細胞(リンパ球、単球などを含む血球細胞)は、中枢神経系の炎症や変性と関連があると考えられています。健常者群および多発性硬化症(multiple sclerosis : MS)患者群脳脊髄液中の細胞のscRNA-seqにより取得したデータの2群間比較解析により、各単核細胞の細胞割合と遺伝子発現における変化(統計的有意差)の検出を行いました。
細胞のクラスタリング結果(19,058 cells, 2群、n=5, UMAP)
細胞のクラスタリング結果(19,058 cells, 2群、n=5, UMAP)
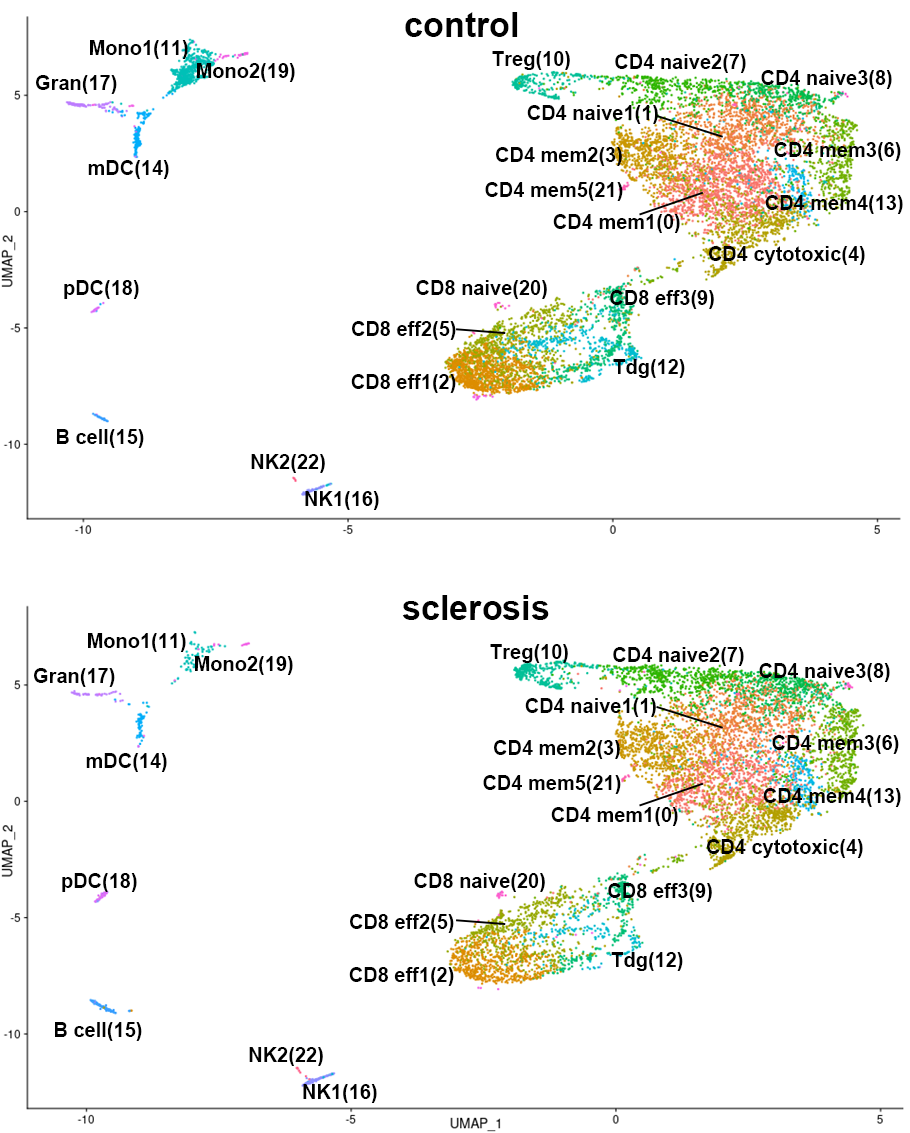

Batch effect(実験間誤差)補正後に発現プロファイルを統合し、クラスタリング結果をUMAPにより二次元上にプロットしました。健常者群(n=5, 10,386 cells)および多発性硬化症群(n=5, 8,672 cells)由来の細胞を群ごとに分けて表示しました(上図:健常者、下図:多発性硬化症患者)。細胞タイプ名に続く( )内の数字はクラスタ番号を示します。
各クラスタに特徴的な遺伝子発現(Dot plot)
各クラスタに特徴的な遺伝子発現(Dot plot)
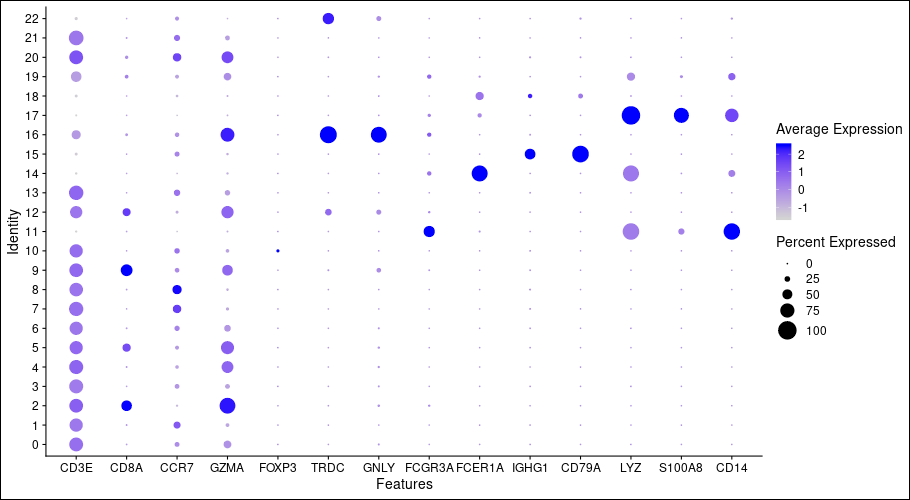

各クラスタに特徴的な遺伝子をドットプロットを用いて可視化しました。縦軸は各クラスタ、横軸は遺伝子に対応しています。発現量をドットの色の濃淡で、発現している細胞の割合をドットの大きさで示しました。
CD4+ memory細胞サブクラスタの発現変動遺伝子(Heatmap)
CD4+ memory細胞サブクラスタの発現変動遺伝子(Heatmap)
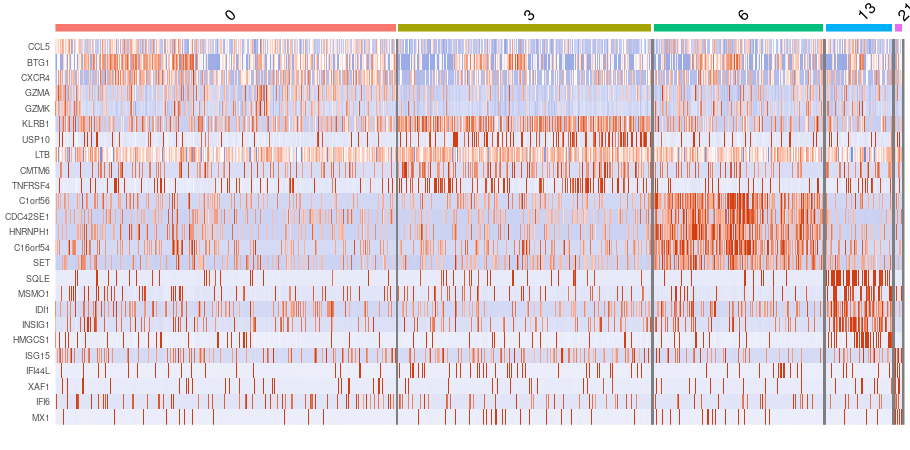

CD4+ memory細胞は5つのサブクラスタ(0, 3, 6, 13, 21)に分類されました。 各クラスタに特徴的な遺伝子発現をヒートマップを用いて可視化しました。縦軸は遺伝子に、横軸は各クラスタに対応しています。発現量が多い遺伝子は赤色、少ないものは青色で示しました。
検体毎の各クラスタの構成細胞数(抜粋)
検体毎の各クラスタの構成細胞数(抜粋)
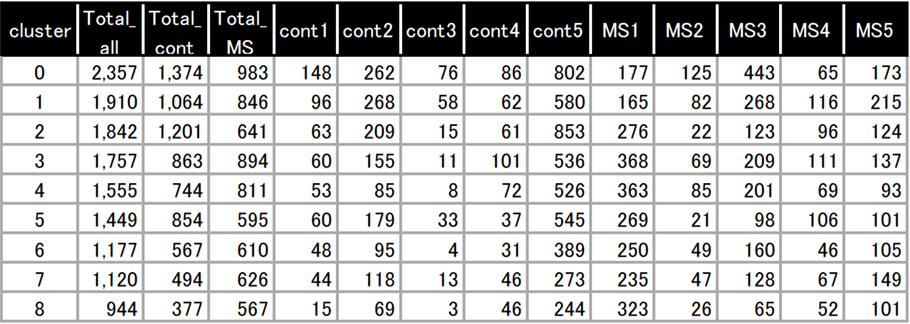

各クラスタに含まれる細胞数を検体毎に算出した結果を表に示しました。
各クラスタの構成細胞割合の比較(抜粋)
各クラスタの構成細胞割合の比較(抜粋)
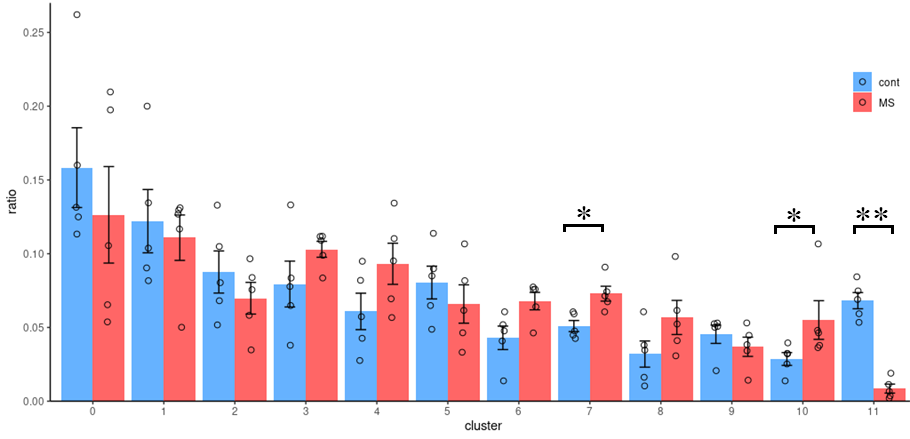

各クラスタに含まれる細胞の割合を検体毎に算出し、健常者群(cont)および多発性硬化症群(MS)に分けてグラフに示しました。縦軸は細胞の割合、横軸は各クラスタを示しています。検体毎の割合を白抜きの円で表示し、各群の平均値(±SEM)を棒グラフで示しました。群間で有意差検定を実施した結果を、*p<0.05, **p<0.01で示しました。
多発性硬化症により発現変動する遺伝子の発現分布
多発性硬化症により発現変動する遺伝子の発現分布
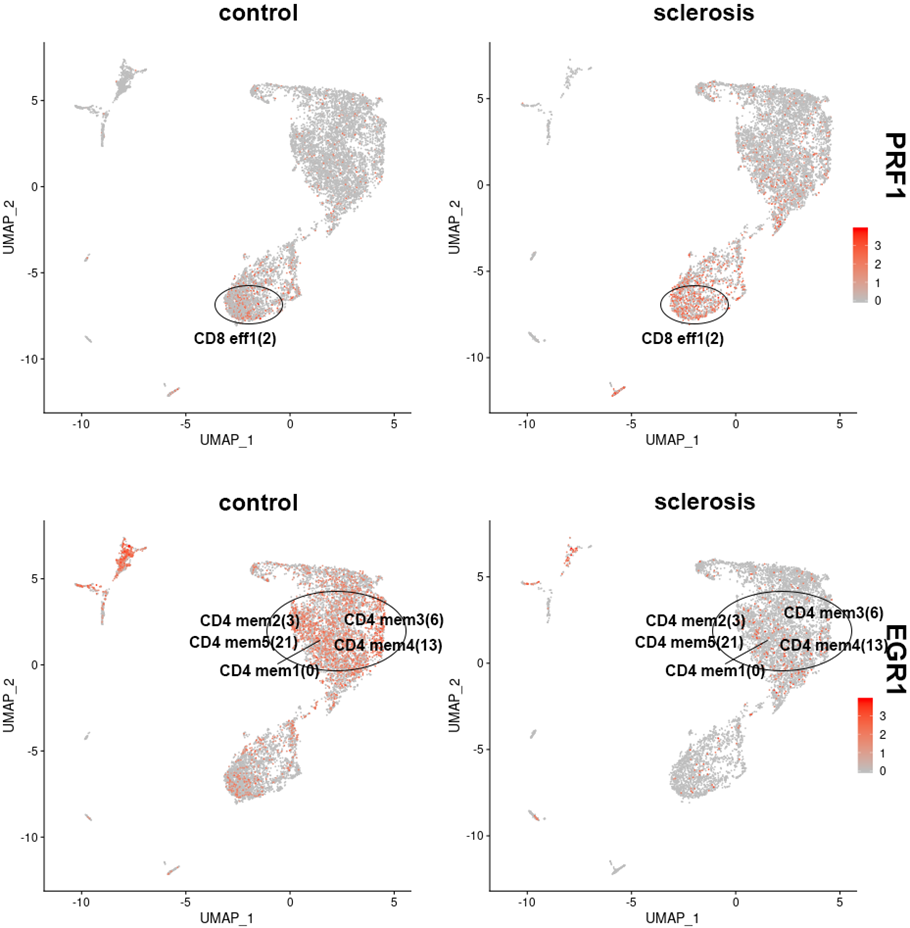

多発性硬化症により発現変動した遺伝子をクラスタ毎に抽出し、その発現分布を二次元上にプロットしました。上段のCD8+ effector 1細胞で発現増加したPerforin-1(PRF1)や、下段のCD4+ memory細胞で発現減少したEarly growth response protein 1(EGR1)を例として示しました。
多発性硬化症により発現変動する遺伝子の発現レベル(Violin plot)
多発性硬化症により発現変動する遺伝子の発現レベル(Violin plot)
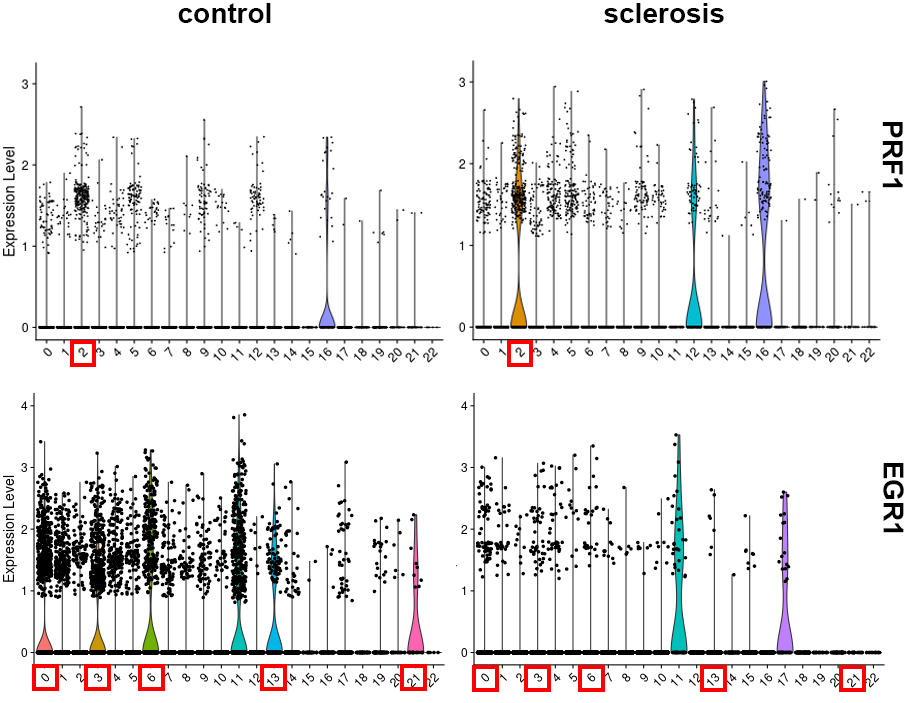

各クラスタにおける遺伝子発現量の変化をバイオリンプロットで図示しました。上段に赤枠で示したCD8+ effector 1(クラスタ2)に特徴的なPRF1 の発現増加や、下段に赤枠で示したCD4+ memory(クラスタ0, 3, 6, 13, 21)に特徴的なEGR1の発現減少が示されました。
解析例3 遺伝子ノックアウトにより異常を生じる少数細胞の発現変動遺伝子抽出
遺伝子Xのノックアウトによって異常が見られる部位になる領域を、野生型(WT)およびノックアウト(KO)マウス胚より取り出しSingle cell RNA sequenceを行ったデータを解析しました。細胞亜集団の検出、細胞タイプ推定、WTとKOにおける発現変動遺伝子(DEG)の抽出を行いました。
細胞のクラスタリング結果(4,088 cells, 2群、n=1, t-SNE)
細胞のクラスタリング結果(4,088 cells, 2群、n=1, t-SNE)

各種遺伝子発現量等のバイアス補正後、実験間誤差(Batch effect)を排除しながら発現プロファイルの次元削減を行い、クラスタリング結果をt-SNE*5により可視化しました。左図はWT、KO毎に、右図はクラスタ毎に図示しました。
各クラスタの細胞タイプ推定結果
各クラスタの細胞タイプ推定結果
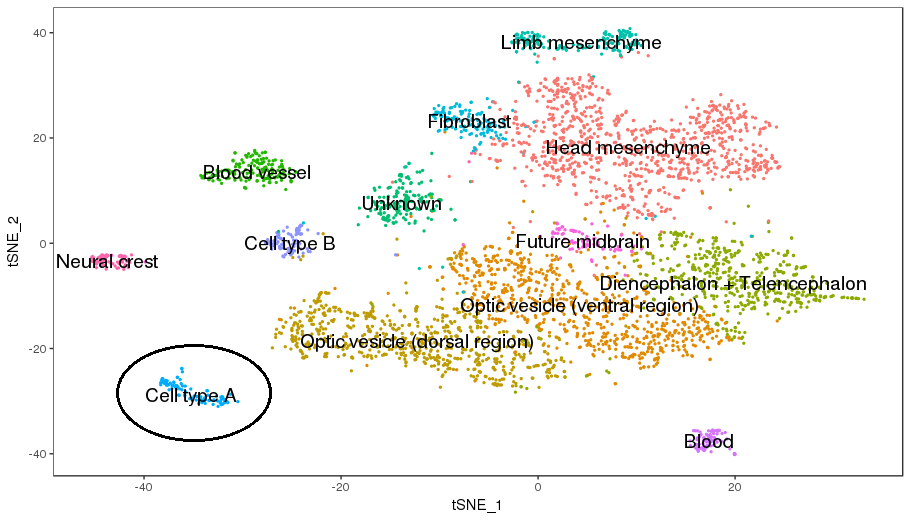
発現変動遺伝子、マーカー遺伝子、GO解析等の機能解析結果より総合的に判断し、各クラスタの細胞タイプを推定し図示しました。Cell type AがKOマウスの表現型の異常に関与すると推定された細胞タイプです。
野生型とノックアウトにおける発現変動遺伝子の発現分布の一例
野生型とノックアウトにおける発現変動遺伝子の発現分布の一例

WTとKOにおいて、発現変動した遺伝子の一部を図示しました。ほとんどの細胞タイプで大きな変化は見られませんが、Cell type Aでは、複数の遺伝子に顕著な発現変動が確認できました。これらの発現変動遺伝子の一部はKOマウスの表現型に深く関わっている可能性が考えられます。この様にscRNA-seq解析では、細胞集団のわずか数%の細胞でしか起こっていない変化を鋭敏に検出することが可能です。
解析例4 がん組織浸潤T細胞に特徴的なサブタイプの同定
末梢血、正常組織、肝腫瘍組織より採取したT細胞から、 FACSにより分取したCD3+/CD8+ T細胞と、CD3+/CD4+ T細胞のscRNA-seqデータを使用して4,721細胞のクラスタリングを行いました。
CD8+ T細胞のクラスタリング結果(1,668/4,721 cells, 5群、n=1, t-SNE)
CD8+ T細胞のクラスタリング結果(1,668/4,721 cells, 5群、n=1, t-SNE)
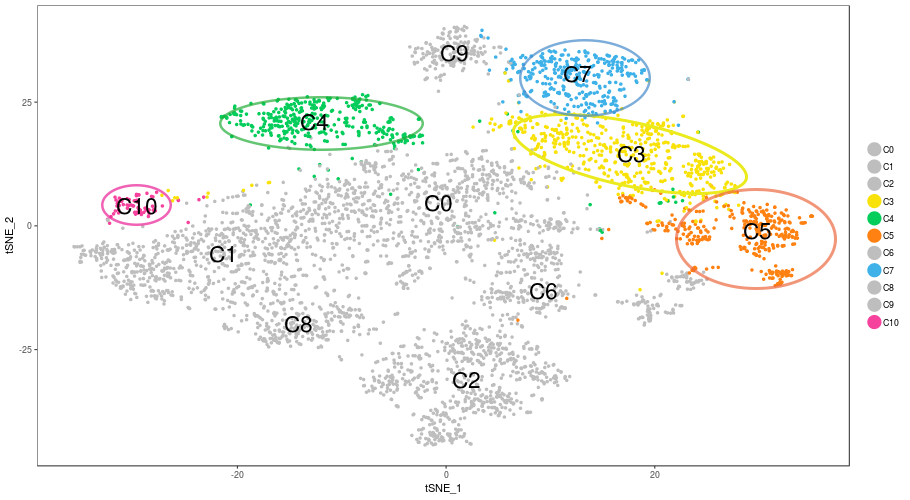
PCA(主成分分析)により発現プロファイルの次元削減を行い、クラスタリング結果をt-SNEにより可視化しました。T細胞(total 4,721 cells)は11のクラスタに分類され、CD8+T細胞は5クラスタ(C3,C4,C5,C7,C10、total 1,668 cells)に分類されました。
遺伝子発現分布
CD8A GZMA遺伝子発現分布
CD8A GZMA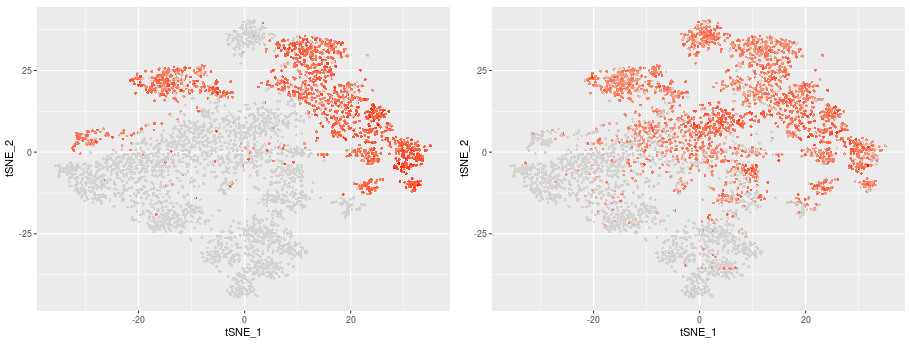
CD8Aを発現する細胞障害性T細胞(C3,C4,C5,C7,C10)は大部分がGZMA (Granzyme A)を発現していました。
各クラスタに特徴的な遺伝子発現ヒートマップ(Heatmap)
各クラスタに特徴的な遺伝子発現ヒートマップ(Heatmap)

各クラスタに特徴的な遺伝子の発現をヒートマップで一覧表示しました。C5では、疲弊した(exhausted)T細胞に特徴的に発現するHAVCR2(TIM-3), TNFRSF9, PDCD1(PD-1)などが発現していました。この様にして、細胞集団の性質を知らずにクラスタリング(unsupervised clustering)した後で、その細胞の性質を解析出来ます。さらに、由来組織を色分け表示(図上部、末梢血:緑、正常組織:青、腫瘍組織:赤)すると、C5のほとんどは腫瘍組織由来でした。
細胞の特徴のスコアリング表示(Signature Scoring)
細胞の特徴のスコアリング表示(Signature Scoring)
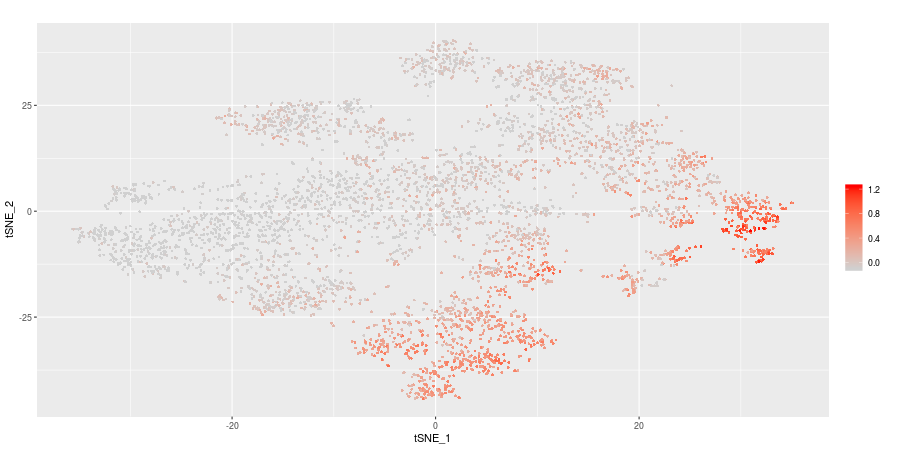
疲弊したT細胞に特徴的な遺伝子群(Zheng et al.)の発現量と、ランダムに選択した遺伝子群の発現量の差分をスコアリングして可視化しました。スコアが高い(赤色で表示)細胞ほど、疲弊したT細胞の特徴を有していると考えられます。C5が最も高いスコアを示しました。
由来組織ごとのCD8+ T細胞構成比
由来組織ごとのCD8+ T細胞構成比

由来組織ごとに各クラスタの構成比率を見ると、C5は末梢血(PB)や正常組織(Normal)にはほとんど見られず、腫瘍組織(Tumor)浸潤T細胞に多く含まれていました。この様に、細胞の分類基準や組成が分かっていない状態で細胞を分類し、解析対象(病態モデルやノックアウトなど)に特徴的なクラスタを見出すことが出来ます。
CD4+ T細胞のクラスタリング結果(3,053/4,721 cells, 5群、n=1, t-SNE)
CD4+ T細胞のクラスタリング結果(3,053/4,721 cells, 5群、n=1, t-SNE)
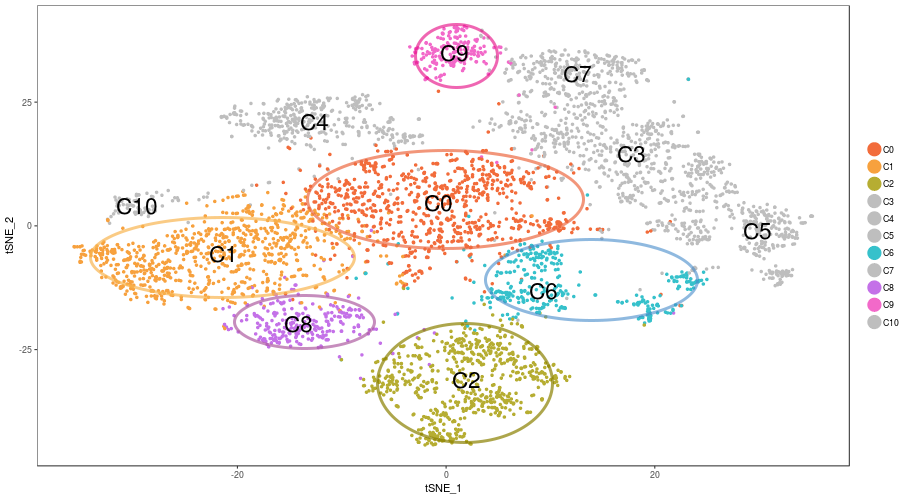
11クラスタに分類されたT細胞 (total 4,721 cells)のうち、CD4+T細胞は6クラスタ(C0, C1, C2, C6, C8, C9、total 3,053 cells)に分類されました。
遺伝子発現分布
CD4 FOXP3遺伝子発現分布
CD4 FOXP3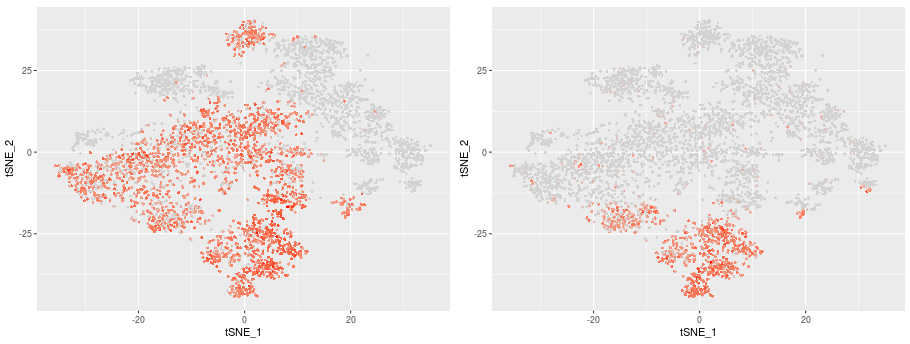
CD4を発現するT細胞(C0,C1,C2,C6,C8,C9)のうち、C2には制御性T細胞(Treg)で特徴的に発現するFOXP3の発現が見られました。
Volcano plotによるC2の発現変動遺伝子の可視化
Volcano plotによるC2の発現変動遺伝子の可視化

FOXP3を発現しているC2とそれ以外のCD4+T細胞の遺伝子発現を比較し、発現変動比と有意確率をVolcano plotで図示しました。C2は制御性T細胞に特徴的なFOXP3, TNFRSF18, TNFRSF9などの発現量が有意に高い(グラフ右上)ことが示されました。
由来組織ごとのCD4+ T細胞構成比
由来組織ごとのCD4+ T細胞構成比

由来組織ごとにCD4+ T細胞の構成比率を見ると、末梢血由来に多いC1とC8は組織中には少なく、また、腫瘍組織由来では正常組織由来と比べて、免疫応答を抑制する制御性T細胞(Treg, C2)の割合が顕著に増加していました。
解析例5 視覚野神経細胞において視覚刺激により発現変化した遺伝子の網羅的検出
暗所飼育後に光刺激を与えたマウスの0時間後と4時間後の視覚野に由来する細胞のSingle cell RNA-seqデータを用いて、各細胞タイプにおける発現変動遺伝子の検出を行いました。
細胞のクラスタリング結果(14,760 cells, 2群、n=3, UMAP)
細胞のクラスタリング結果(14,760 cells, 2群、n=3, UMAP)
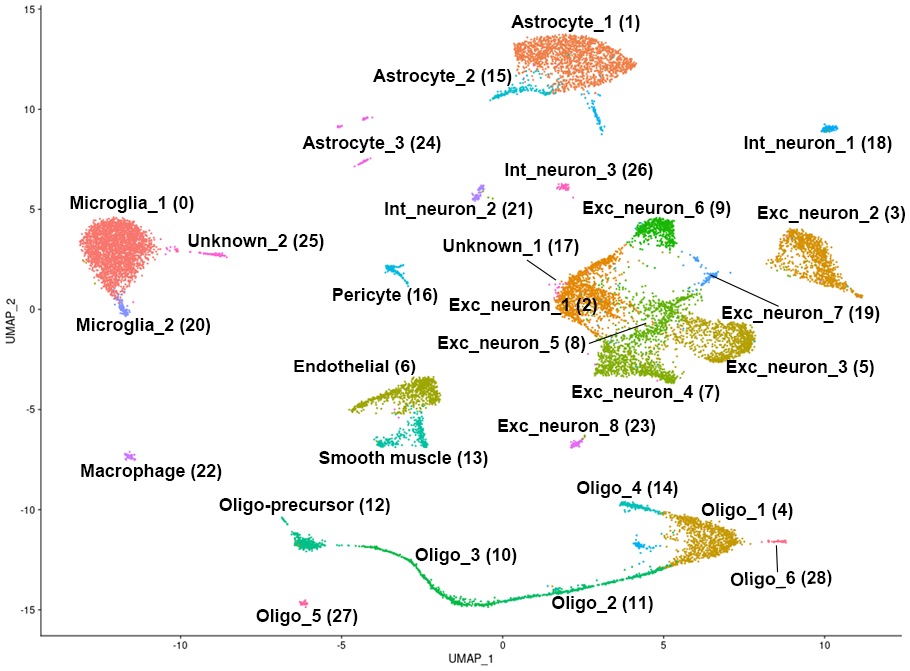

光刺激0時間後および4時間後の視覚野由来細胞のSingle cell RNA-seqデータをbatch effect correction (実験間誤差補正)後に統合し、クラスタリング結果をUMAPによりプロットしました。細胞タイプ名に続く( )内の数字はクラスタ番号を示します。
各クラスタの発現変動遺伝子(Heatmap)
各クラスタの発現変動遺伝子(Heatmap)
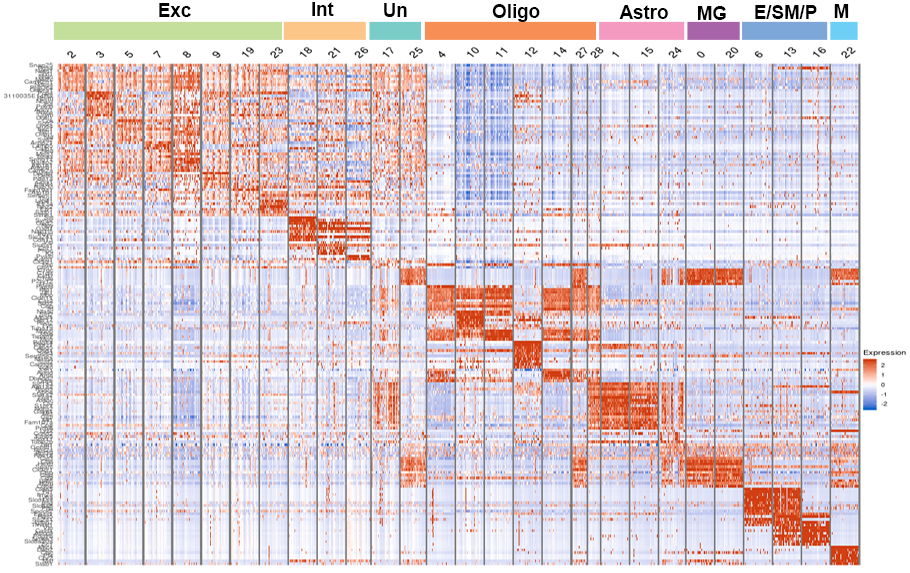

各クラスタに特徴的な遺伝子の発現量をヒートマップを用いて可視化しました。各クラスタの細胞数を上限100としてダウンサンプリングした結果を示します。上部のカラーバーは、細胞タイプを示します。
神経細胞における各遺伝子の発現レベル(Violin plot)
神経細胞における各遺伝子の発現レベル(Violin plot)
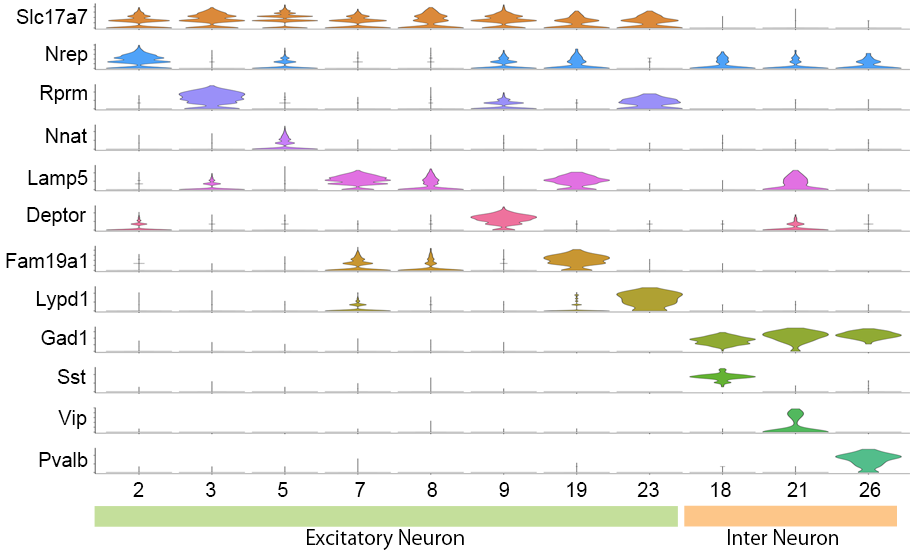

神経細胞と推定されたクラスタについて、各遺伝子の発現レベルをバイオリンプロットを用いて可視化しました。Excitatory neuronにはSlc17a7、Inter neuronにはGad1の特異的な発現が見られました。Inter neuronと推定されたクラスタでは、クラスタ毎に異なる神経ペプチドなど(Sst, Vip, Pvalb)の発現が見られました。
各クラスタの細胞分布および割合の比較
各クラスタの細胞分布および割合の比較
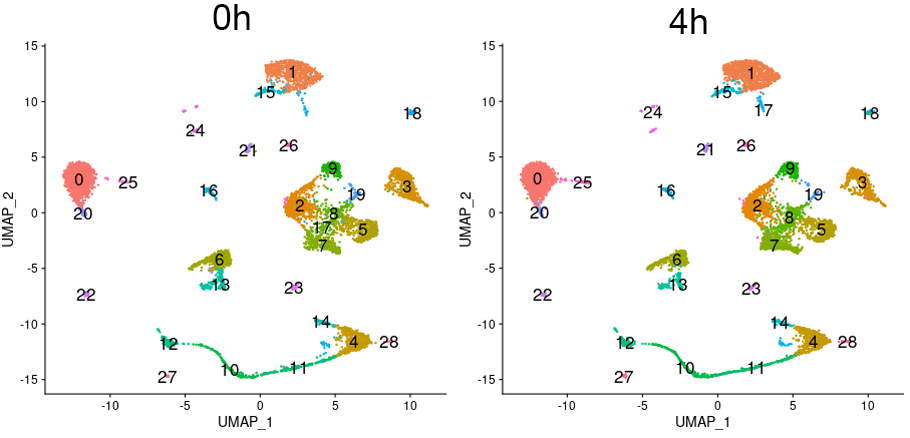

クラスタリング結果を、光刺激0時間後および4時間後の細胞に分けて表示しました。各クラスタの細胞構成比や分布に大きな変化は見られませんでした。図中の数字はクラスタ番号を示します。
各細胞タイプにおける発現変動遺伝子の可視化(Volcano plot)
各細胞タイプにおける発現変動遺伝子の可視化(Volcano plot)
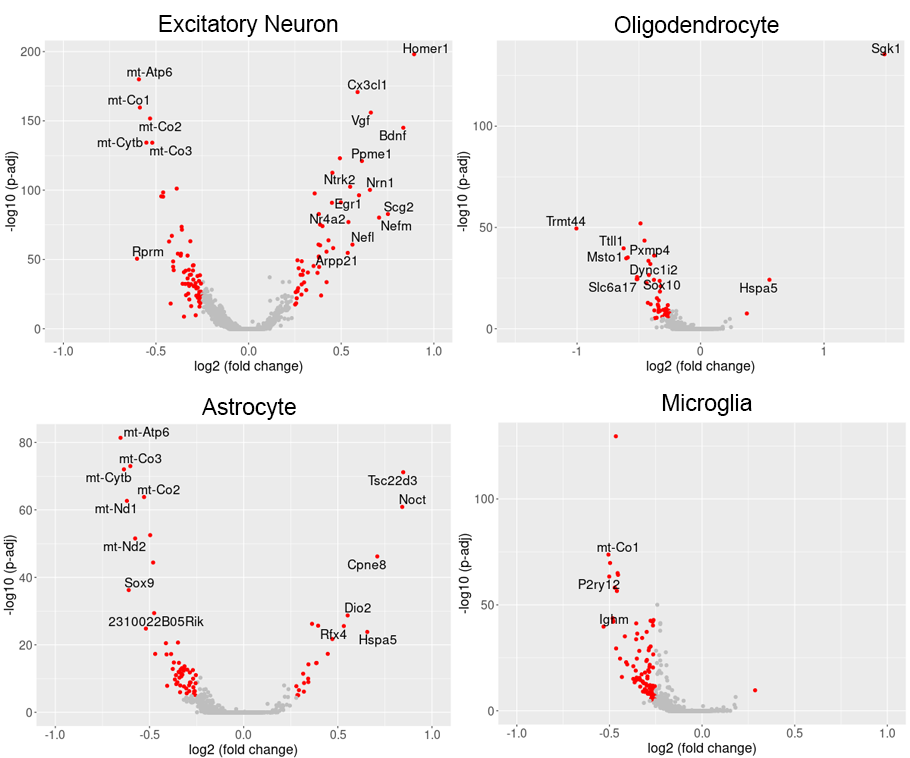

細胞タイプごとの平均遺伝子発現量を、光刺激0時間後と4時間後で比較し、Volcano plotにより各遺伝子の発現変動比と統計的有意確率を表示しました。細胞タイプごとに固有の発現変動遺伝子が検出されました。
各細胞タイプにおける初期応答転写因子の発現変化の可視化
各細胞タイプにおける初期応答転写因子の発現変化の可視化
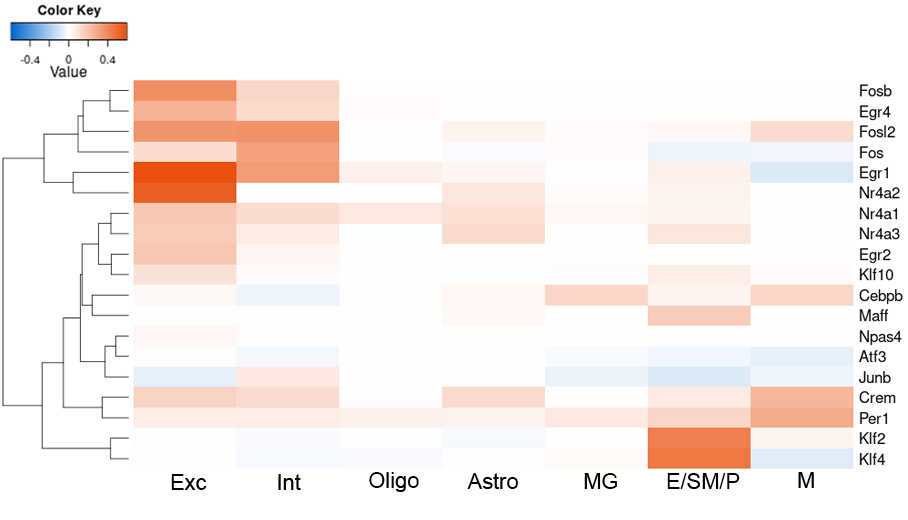

培養神経細胞を用いた実験などにより、神経活動により発現が変化することが知られている初期応答転写因子の細胞タイプごとの発現変化(log2FC)をヒートマップ表示しました。
解析例6 抗原提示細胞の分化経路推定(trajectory analysis)
ヒト単球(Monocyte)を適切な培養条件で分化誘導すると、CD1+樹状細胞(Dendritic cell)やCD16+マクロファージ(Macrophage)に分化することが知られています(Krutzik et al.)。
がん患者腹水から樹状細胞としてCD1c+/CD16-細胞、マクロファージとしてCD1c-/CD16+細胞、末梢血中より単球としてCD14+細胞をFACSにより分取し、Single cell RNA-seqを行ったデータを使用して解析を行いました。
細胞のクラスタリング結果(4,002 cells, 3群、t-SNE)
細胞のクラスタリング結果(4,002 cells, 3群、t-SNE)
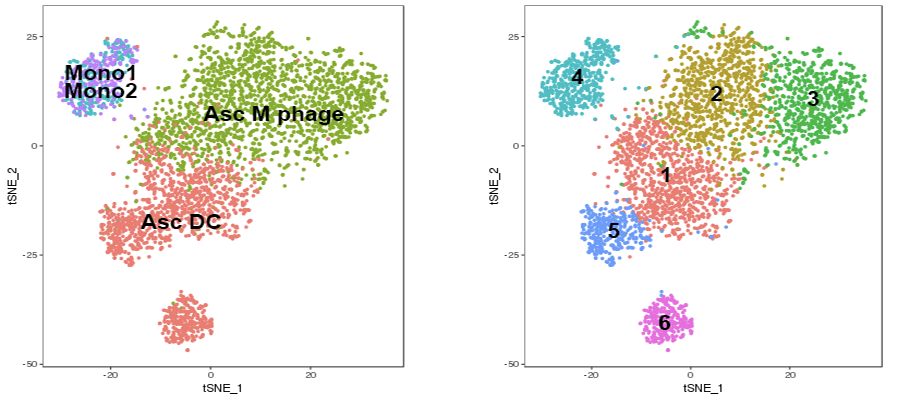
PCAによる次元削減後に細胞のクラスタリングを行い、t-SNEにより二次元上に可視化しました。右図はクラスタごと、左図はFACS分取した由来細胞タイプごとに色分け表示しました。腹水より採取された樹状細胞およびマクロファージは単球由来であることが示されており、これらの細胞の発現プロファイルを用いて分化経路推定を行いました。
| 1: mo-derived dendritic cell 1 | 2: macrophage 1 |
| 3: macrophage 2 | 4: monocyte |
| 5: mo-derived dendritic cell 2 | 6: end-stage mo-derived dendritic cell |

PCAによる次元削減後に細胞のクラスタリングを行い、t-SNEにより二次元上に可視化しました。右図はクラスタごと、左図はFACS分取した由来細胞種ごとに色分け表示しました。腹水より採取された樹状細胞およびマクロファージは単球由来であることが示されており、これらの細胞の発現プロファイルを用いて分化経路推定を行いました。
1: mo-derived dendritic cell 1
2: macrophage 1
3: macrophage 2
4: monocyte
5: mo-derived dendritic cell 2
6: end-stage mo-derived dendritic cell
分化経路推定(trajectory analysis)
cluster pseudotime分化経路推定(trajectory analysis)
cluster pseudotime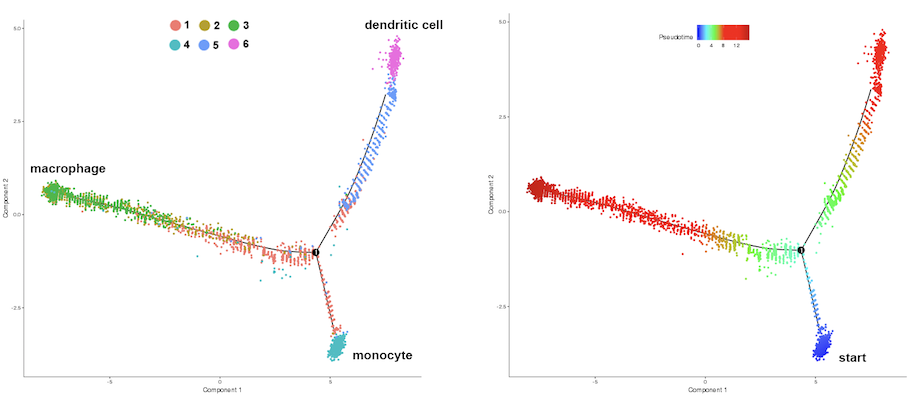
特徴的な遺伝子発現情報を基に発現プロファイルの次元削減を行い、二次元上に分化経路を再構築しました。再構築した経路に沿って各細胞を配置し、個々の細胞の擬時間(pseudotime)を算出しました。左図はクラスタごと、右図は擬時間に従って細胞を色分け表示しました。
擬時間経過による遺伝子発現変化(Heatmap)
macrophage monocyte dendritic cell擬時間経過による遺伝子発現変化(Heatmap)
macrophage monocyte dendritic cell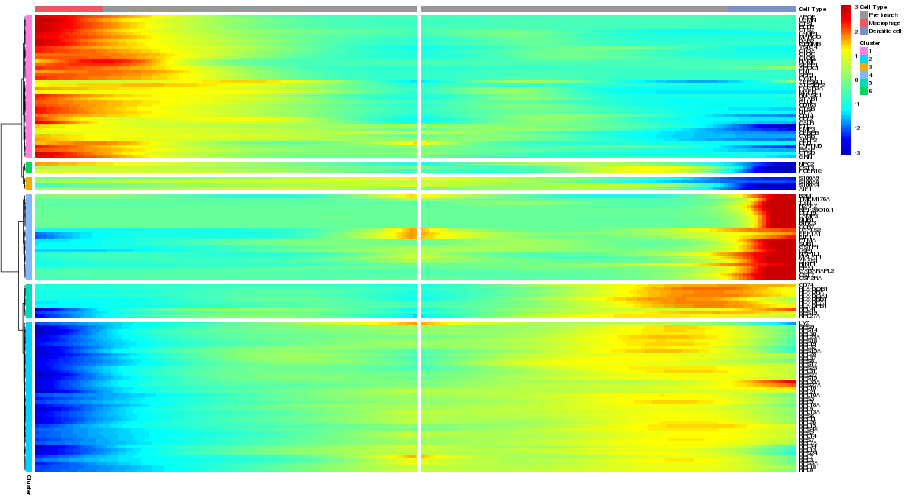
擬時間変化に伴って発現量が変動した遺伝子を網羅的に抽出し、各遺伝子の発現量をヒートマップ表示しました。ヒートマップ中央を分化(および擬時間)の始点(monocyte)として、左側がmacrophage、右側がdendritic cellに分化する方向に擬時間経過を表示しました。
擬時間経過による各遺伝子の発現変化
Monocyte / Macrophage markers Dendritic cell markers擬時間経過による各遺伝子の発現変化
Monocyte / Macrophage markers Dendritic cell markers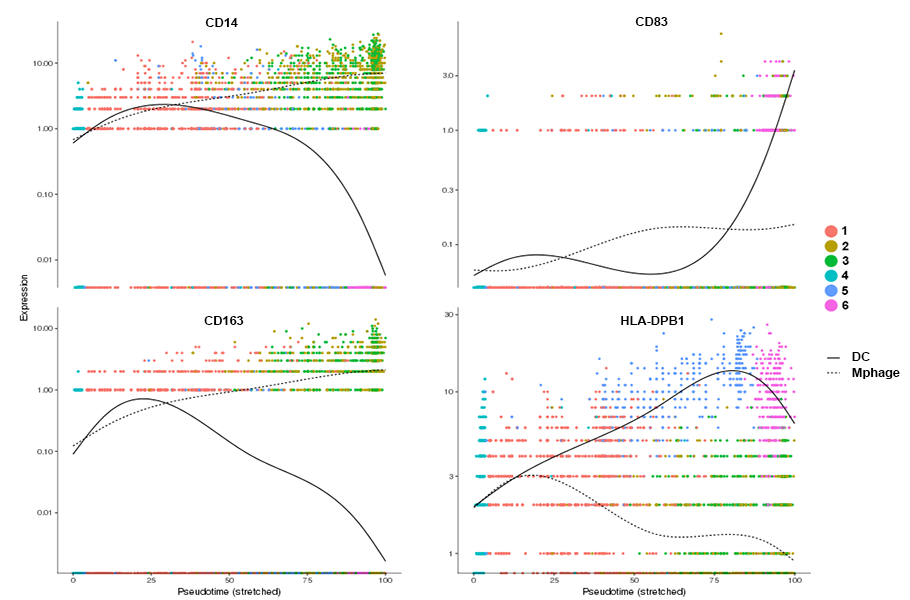
擬時間経過に伴って変化する遺伝子の中から、既知のdendritic cellおよびmonocyte / macrophageマーカー遺伝子を抽出し、擬時間経過に伴う発現量変化をプロットしました。縦軸は発現量、横軸は擬時間経過、着色された各点は個々の細胞を示しています。擬時間は、始点を0、それぞれの経路の終点を100として表示しました。実線がdendritic cell側、点線がmacrophage側に分岐した細胞での発現量変化を示しています。
擬時間経過による遺伝子発現変化(分化経路上にプロット)
Monocyte / Macrophage marker Dendritic cell marker擬時間経過による遺伝子発現変化(分化経路上にプロット)
Monocyte / Macrophage marker Dendritic cell marker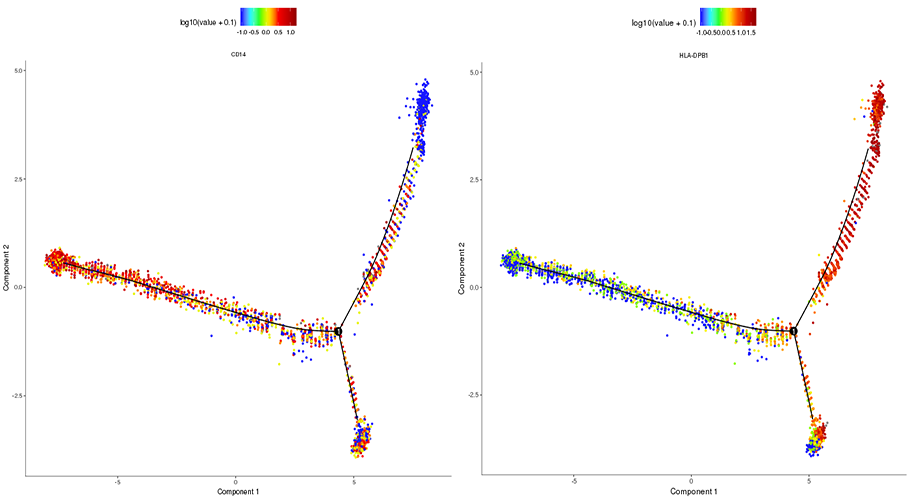
分化経路推定を行った二次元プロット上に、dendritic cellおよびmonocyte / macrophageマーカー遺伝子の発現量変化をプロットしました。各マーカー遺伝子の擬時間経過に伴う発現増加が確認できます。
解析例7 RNA velocity解析による細胞動態の推定
げっ歯類の切歯は生涯伸び続けることが知られています。これはげっ歯類の切歯の根元に存在する幹細胞が、歯のエナメル質を形成するエナメル芽細胞を継続的に供給していることによります。spliced RNAとunspliced RNAの割合の違いから細胞状態遷移の向きと大きさを推定する手法であるRNA velocity解析により、切歯基部の細胞動態を解析しました。
細胞のクラスタリング結果(3,296 cells, 1群、n=1, UMAP)
細胞のクラスタリング結果(3,296 cells, 1群、n=1, UMAP)
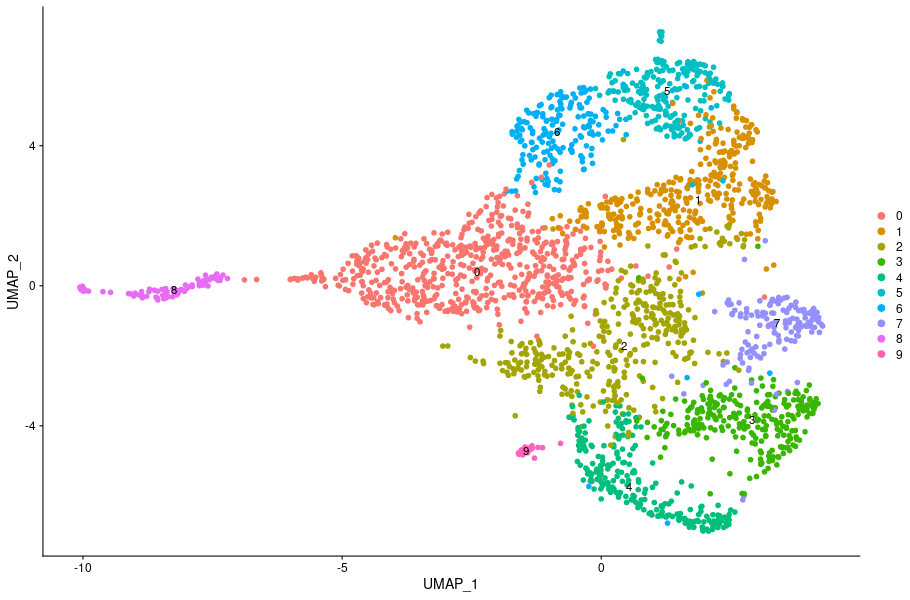
エナメル芽細胞と前駆細胞を含むマウス切歯基部の上皮細胞をクラスタリングしました。PCAにより発現プロファイルの次元削減を行い、クラスタリング結果をUMAPにより二次元上に可視化しました。
3次元プロットはこちらからご覧頂けます。※プロットはドラックして回転、マウスのホイールで拡大、縮小が可能です。読み込みに時間が掛かる場合があります。
各クラスタに特徴的な遺伝子発現分布
各クラスタに特徴的な遺伝子発現分布
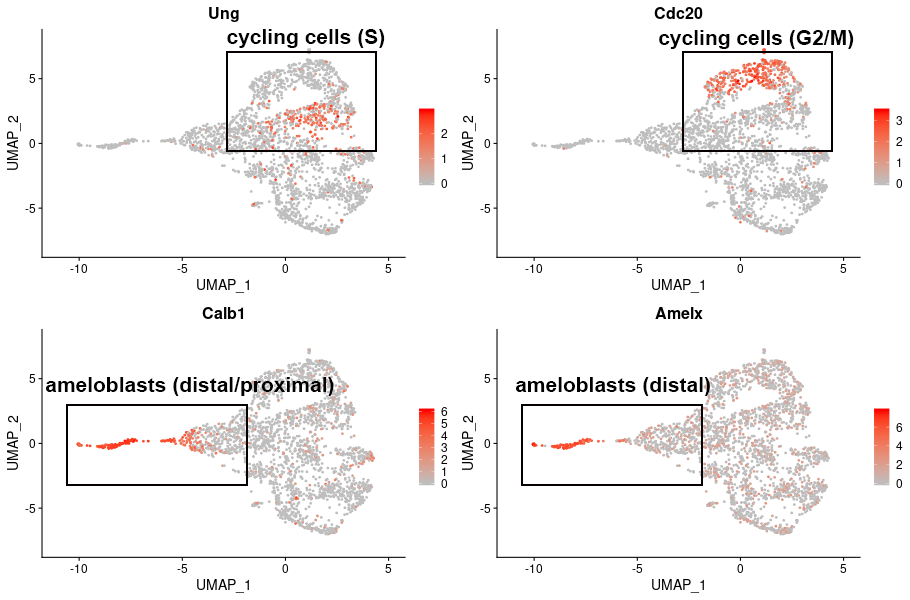
切歯基部の前駆細胞はゆっくりと細胞周期が進行していること、エナメル芽細胞は切歯の近位から遠位に向けて分化が進むことが知られています。S期およびG2/M期に特徴的な遺伝子と、エナメル芽細胞に特徴的な遺伝子の発現分布を示しました。
RNA velocityを用いた細胞動態の可視化
RNA velocityを用いた細胞動態の可視化
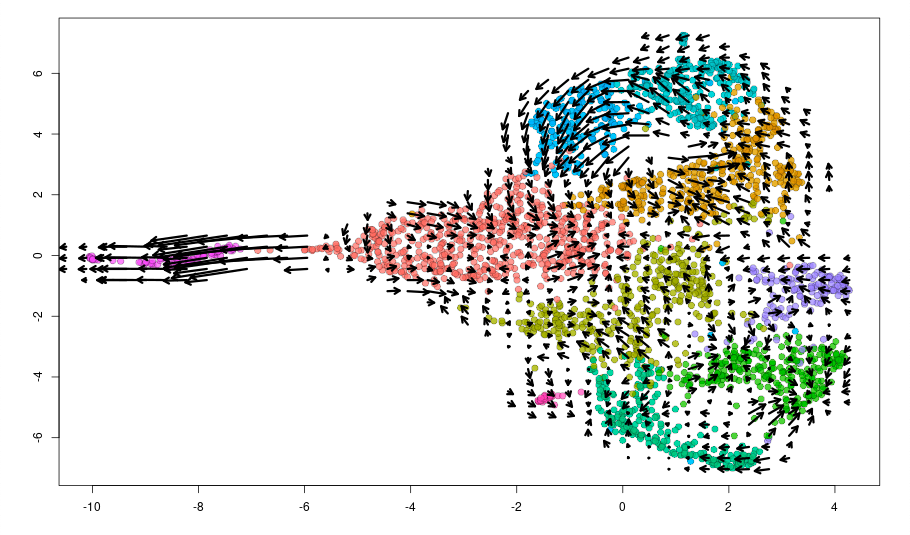
RNA velocity解析により細胞動態を解析しました。矢印は状態変化の方向と大きさを示します。前駆細胞の細胞周期進行と、エナメル芽細胞の分化に伴う状態変化が特徴的に検出されました。
解析例8 エナメル芽細胞分化に伴って発現が変化する遺伝子群の抽出
解析例7でRNA velocity解析により細胞状態が変化する方向が明らかになりました。さらに擬似時間解析(pseudotime解析)により、状態変化に伴って発現が変化する遺伝子群を網羅的に抽出しました。
3次元プロット上での分化経路推定(trajectory analysis)結果
3次元プロット上での分化経路推定(trajectory analysis)結果
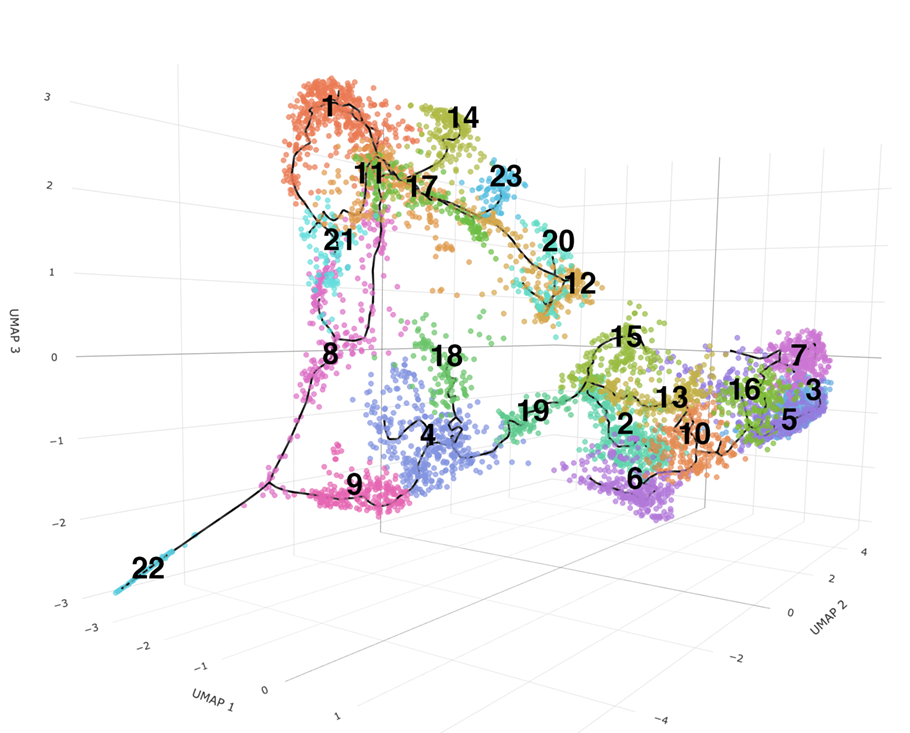

マウス切歯基部由来の上皮細胞をクラスタリングし、経路推定(軌道解析)を行いました。推定された経路を実線(黒)で表示しました。
3次元プロットはこちらからご覧頂けます。※プロットはドラックして回転、マウスのホイールで拡大、縮小が可能です。読み込みに時間が掛かる場合があります。
解析対象経路と擬似時間表示
cluster pseudotime解析対象経路と擬似時間表示
cluster pseudotime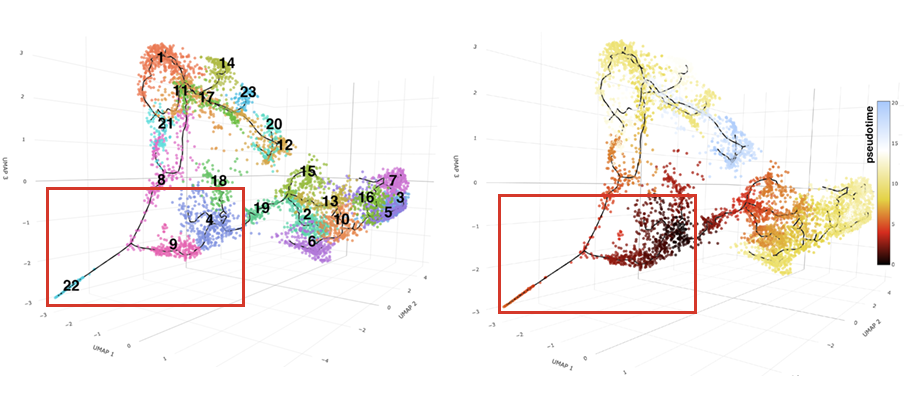

クラスタ4はエナメル芽細胞の前駆細胞、クラスタ22の先端部が分化が進んだエナメル芽細胞と推定されました。クラスタ4から22に至る、4、9、22の分化経路を解析対象としました。クラスタ4を始点として擬似時間を色分け表示しました(右図)。
解析対象クラスタに特徴的な遺伝子群の抽出
解析対象クラスタに特徴的な遺伝子群の抽出
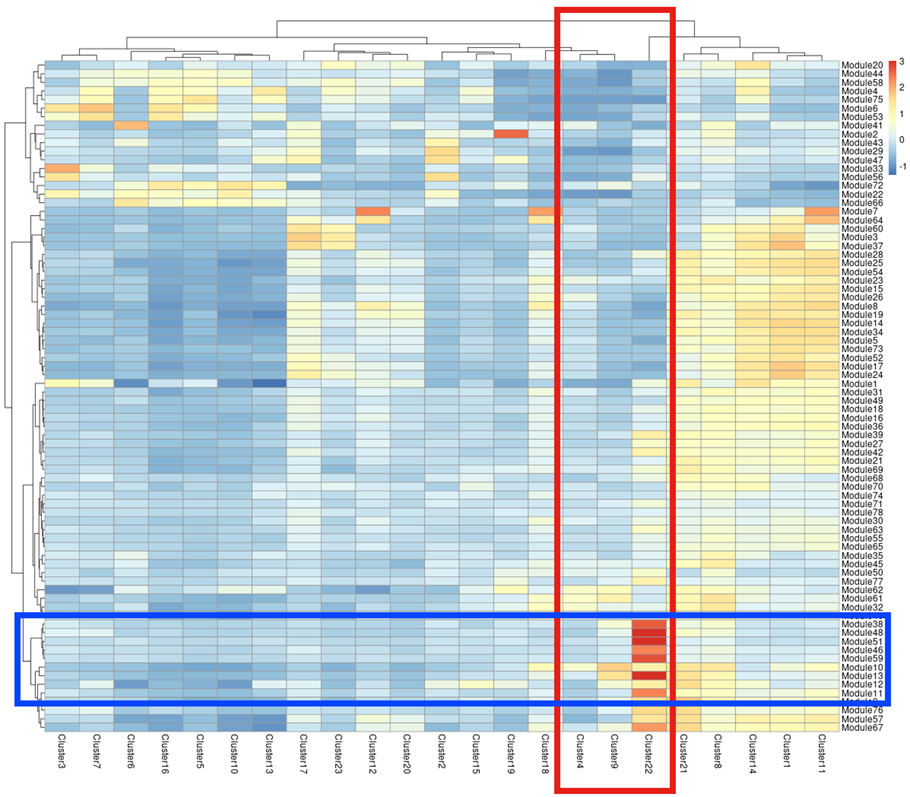

各クラスタに特徴的な遺伝子の発現量をヒートマップで可視化しました。各遺伝子は、発現プロファイルの類似度により、モジュール(遺伝子群)化しました。縦軸は遺伝子群、横軸はクラスタに対応しています。分化経路のクラスタ(4、9、22)を赤枠、最終分化したエナメル芽細胞で発現が増加するモジュールを青枠で示しました。
分化経路に沿って発現上昇した遺伝子の抽出(時系列解析)
分化経路に沿って発現上昇した遺伝子の抽出(時系列解析)
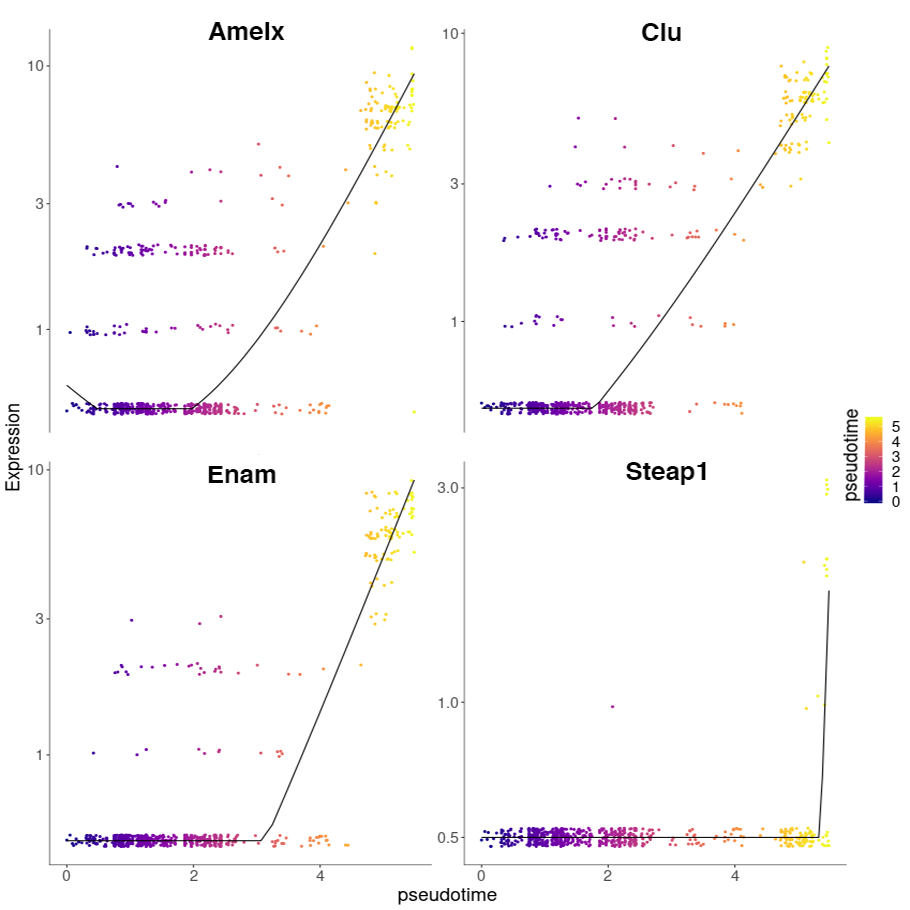

分化の最終段階で発現が上昇した遺伝子モジュールには、分化したエナメル芽細胞で発現することが知られているAmelogenin (Amelx)、Enamelin (Enam)などが含まれていました。グラフの各点は細胞を示しており、解析対象クラスタに含まれる細胞のみを表しています。
分化経路に沿って発現上昇した遺伝子の発現分布と変化
分化経路に沿って発現上昇した遺伝子の発現分布と変化
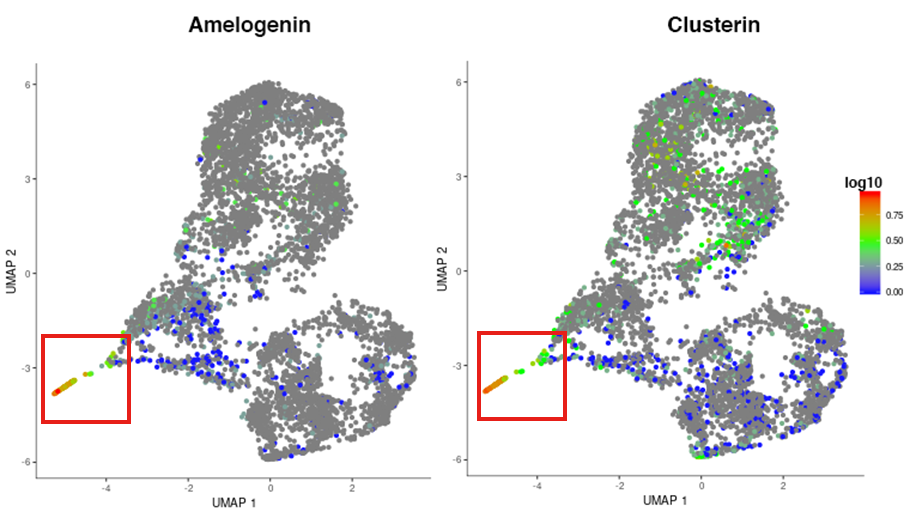

エナメル芽細胞のマーカーであるAmelogenin と、今回の解析で抽出したClusterin (Clu)遺伝子の発現を二次元プロット上で可視化しました。
解析例9 誘導法の違いによる人工多能性幹細胞(iPS細胞)誘導経路の検証
分化した体細胞に、多能性維持に必要な遺伝子群を導入することなどにより、人工多能性幹細胞(iPS細胞)を誘導できる体細胞リプログラミングが知られています。Mouse embryonic fibroblast (MEF)細胞にOct4, Sox2, Klf4を導入して体細胞リプログラミングを誘導した群(OSK群)と、化学的誘導処理によってリプログラミングを誘導した群(CIP群)のシングルセルトランスクリプトームデータを使用して、多能性幹細胞誘導過程の擬時間解析(pseudotime解析)を行いました。
細胞のクラスタリング結果 (24,000 cells, 2群、各群4時点, UMAP)
細胞のクラスタリング結果 (24,000 cells, 2群、各群4時点, UMAP)

MEF細胞にOct4, Sox2, Klf4を導入して0, 3, 6, 8日後、および、化学的誘導処理を行って0, 3, 6, 9日後に取得されたデータを統合してクラスタリングした結果をUMAPにより二次元上にプロットしました。各検体の細胞数を3,000細胞に統一して解析を行いました。
多能性幹細胞マーカー遺伝子の発現分布
多能性幹細胞マーカー遺伝子の発現分布
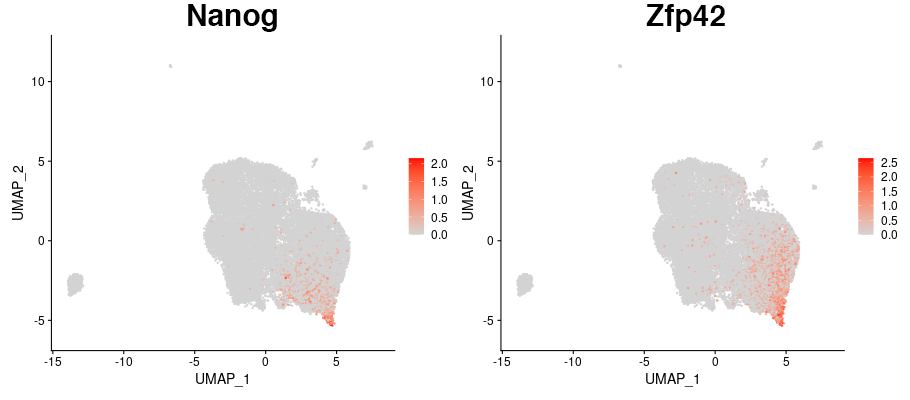

多能性幹細胞のマーカー遺伝子として知られるNanogとZfp42の発現分布を図示しました。一部の共通する細胞集団でNanogとZfp42の発現が見られました。
多能性幹細胞マーカー遺伝子の発現レベル
多能性幹細胞マーカー遺伝子の発現レベル
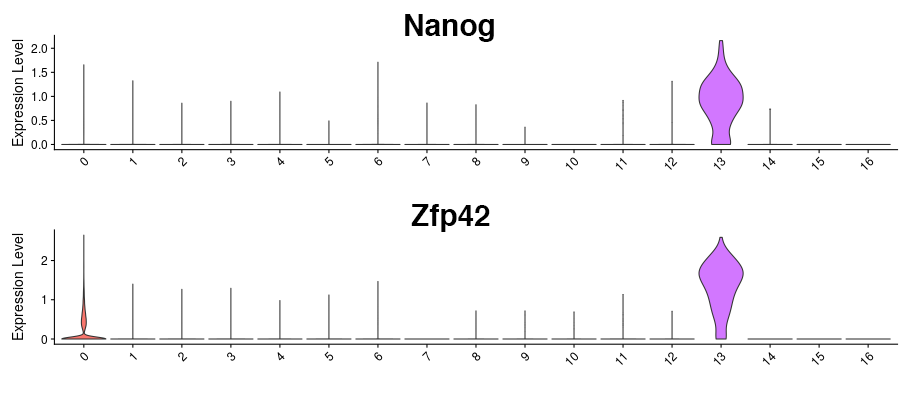

各遺伝子の発現レベルをバイオリンプロットで図示しました。縦軸はそれぞれの遺伝子発現量、横軸は各クラスタを示します。クラスタ13にはNanogおよびZfp42の特徴的な発現が見られ、多能性幹細胞様の細胞と推定されました。
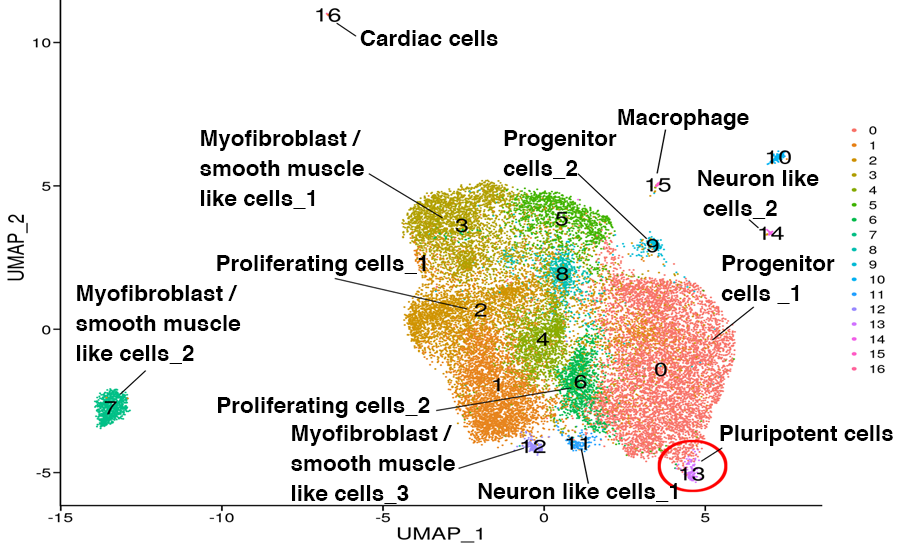
3次元プロット上でのcell lineage推定結果
3次元プロット上でのcell lineage推定結果
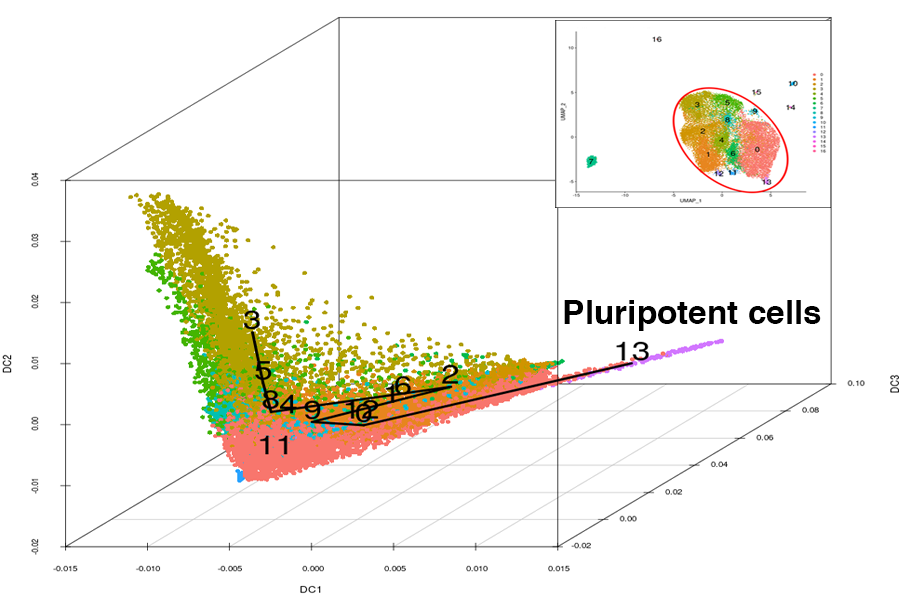

パネル右上に示したクラスタリング結果の赤い円で囲んだクラスタを選択し、diffusion mapにより3次元上にプロットしました。 各クラスタ番号と細胞をプロットした色はパネル右上と対応しています。 3次元プロット上の細胞について、発現プロファイルの類似度からcell lineage(細胞系譜)を推定し、多能性幹細胞を含む細胞系譜を黒い線で表示しました。
3次元プロット上の細胞分布(検体別)
3次元プロット上の細胞分布(検体別)
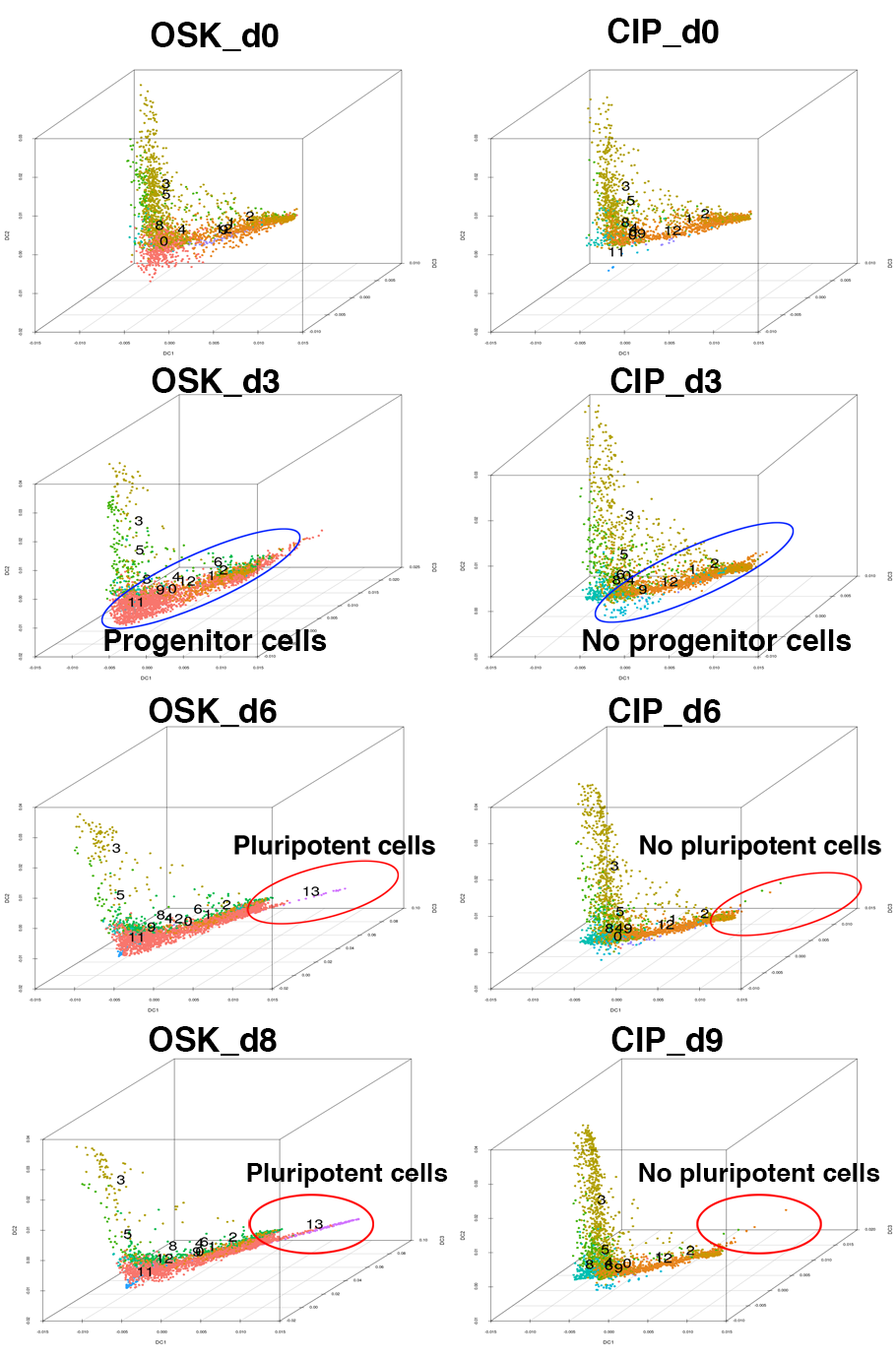

3次元上にプロットした細胞分布を検体別に表示しました。OSK群では時間経過により、まず多能性幹細胞の前駆細胞(青丸内)が誘導され、さらに多能性幹細胞(赤丸内)が誘導されたのに対し、CIP群ではそれらの細胞の誘導は確認されませんでした。
各クラスタの細胞構成比(検体別)
各クラスタの細胞構成比(検体別)
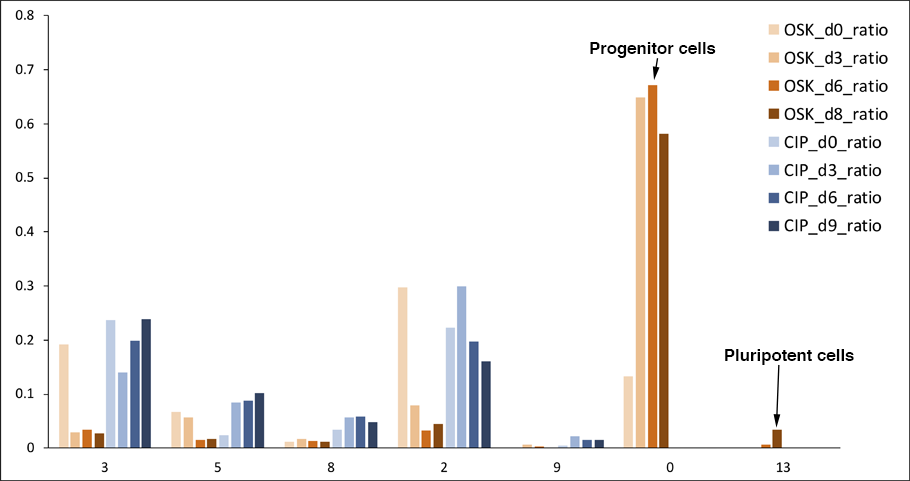

多能性幹細胞に至る系譜として推定された系譜上の各クラスタ(3>5>8>2>9>0>13)の細胞構成比を検体別に表示しました。縦軸は各検体の全細胞数を1とした時の各細胞タイプの割合を示し、横軸はクラスタ番号を示します。OSK群では誘導後の時間経過に伴ってクラスタ0、13に含まれる細胞が増加するのに対し、CIP群では増加が全く見られませんでした。この実験条件下では化学的な刺激による多能性幹細胞の誘導は起こらなかった可能性が考えられました。
多能性誘導経路推定(trajectory analysis)結果
多能性誘導経路推定(trajectory analysis)結果
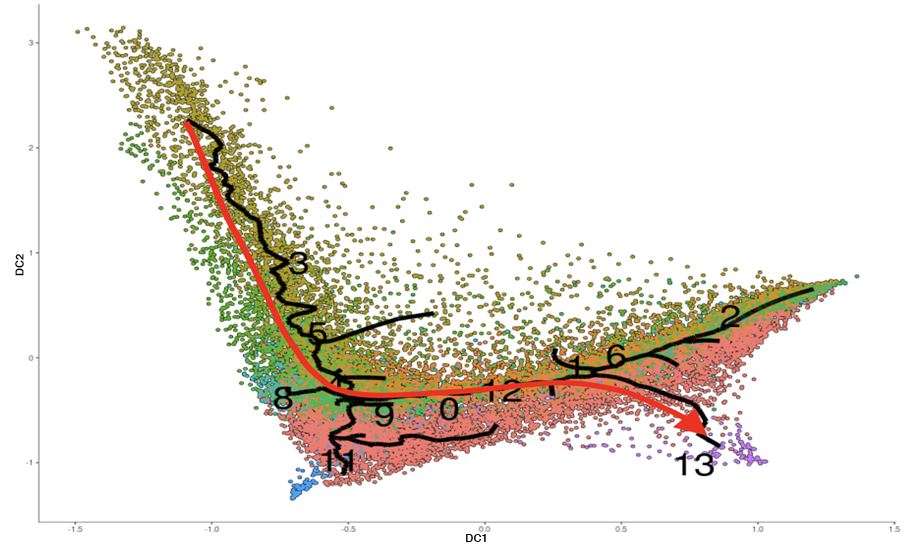

細胞を二次元上にプロットし、多能性誘導経路の推定を行いました。多能性幹細胞に向かう経路を赤色の矢印で示しました。
擬時間経過による遺伝子発現変化(群別)
擬時間経過による遺伝子発現変化(群別)
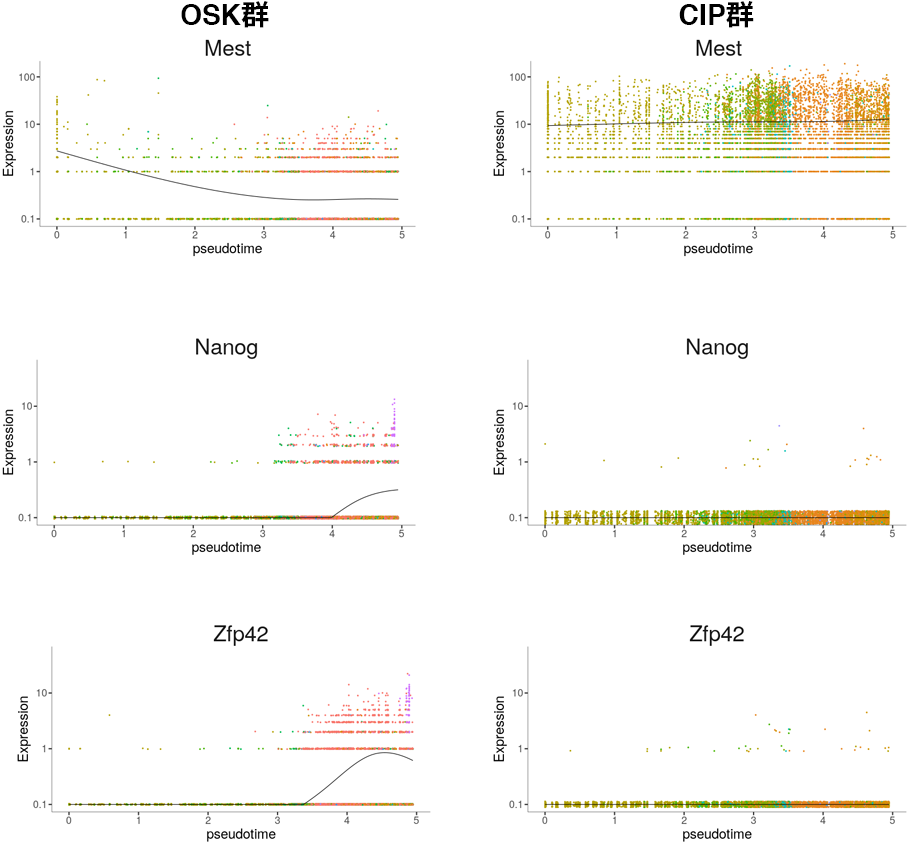

推定した経路上の各細胞の擬時間(pseudotime)と遺伝子発現量より、擬時間経過に伴う遺伝子発現変化を推定し群別にプロットしました。縦軸は遺伝子発現量、横軸は擬時間経過を示し、図中の各点は個々の細胞を示します。OSK群では擬時間経過に従って、中胚葉に特徴的に発現するMest (Mesoderm Specific Transcript)の発現が減少し、多能性幹細胞のマーカー遺伝子として知られるNanogおよびZfp42の発現上昇が見られました。一方、CIP群では擬時間が経過してもMestの発現が維持されており、 NanogおよびZfp42の発現上昇は見られませんでした。この条件下では、化学的な誘導による多能性幹細胞の誘導は見られず、中胚葉様の性質を維持していたと考えられます。
解析例10 疲弊したCAR-T細胞で活性化する転写因子の推定
CAR-T細胞を治療に用いた場合の課題の一つが、T細胞が恒常的に活性化することによってがん細胞を攻撃できなくなったり増殖が抑制されたりするT細胞の疲弊という現象です。CAR-T療法実施後の患者から取得されたCAR-T細胞のシングルセル解析データから、疲弊したT細胞を同定し活性化している転写因子を推定しました。
CAR-T細胞のクラスタリング結果(11,464 cells, 2群、n=3, UMAP)
CAR-T細胞のクラスタリング結果(11,464 cells, 2群、n=3, UMAP)
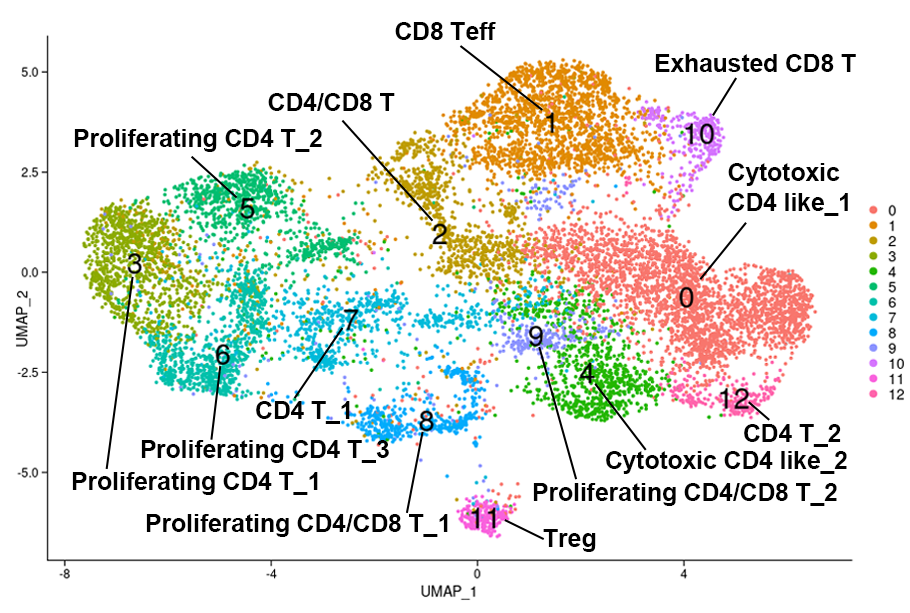

患者に投与したCAR-T細胞(投与群)と、投与7日後に患者末梢血から回収したCAR-T細胞(回収群)のscRNA-seqデータを統合し、クラスタリング結果を二次元上にプロットしました。全細胞のクラスタリング結果からT細胞のみを選択して再クラスタリングを行った結果を示します。
CAR-T細胞のクラスタリング結果(群ごと)
CAR-T細胞のクラスタリング結果(群ごと)
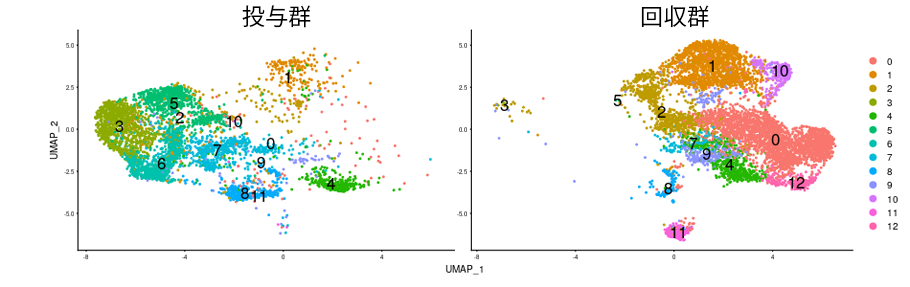

クラスタリング結果を投与群(左)と回収群(右)に分けて表示しました。投与群と回収群ではT細胞集団の構成に顕著な違いが見られました。
T細胞サブセットに特徴的な遺伝子の発現分布
T細胞サブセットに特徴的な遺伝子の発現分布
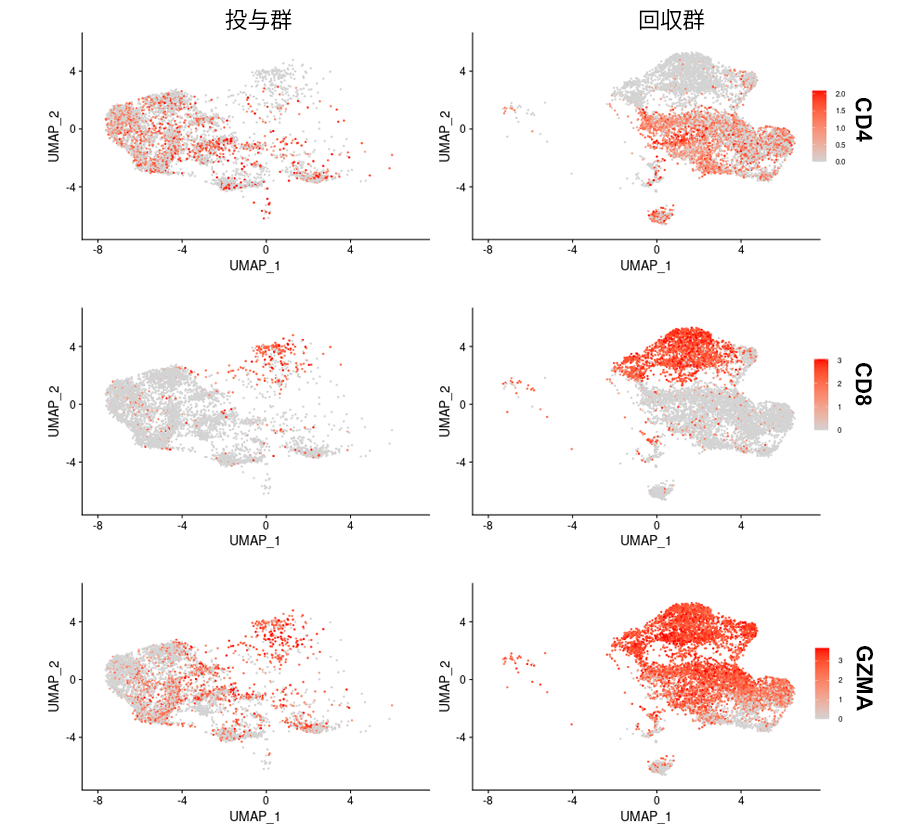

T細胞サブセットに特徴的な遺伝子の発現分布を示しました。投与群(左)に比べ、回収群(右)ではCD8 T細胞の顕著な増加が見られました。さらに回収群では、GZMA遺伝子を発現する細胞障害活性を持つとみられるCD4 T細胞が顕著に増加しており、確かにT細胞サブセットの構成が大きく変化していました。
疲弊したT細胞に特徴的な遺伝子群の発現分布
疲弊したT細胞に特徴的な遺伝子群の発現分布
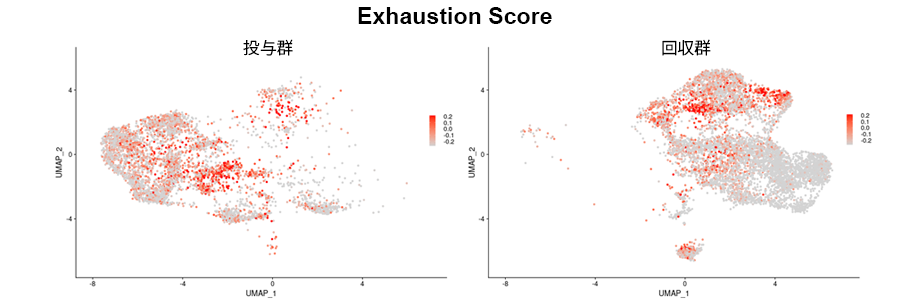

疲弊したT細胞に特徴的な遺伝子群(82遺伝子)の発現をスコアリングし、群ごとに表示しました。スコアが高い細胞を赤色で示しています。回収群では高いスコアを示す疲弊したCD8 T細胞が多く見られました。
各T細胞サブセットで活性化している転写因子の推定
各T細胞サブセットで活性化している転写因子の推定
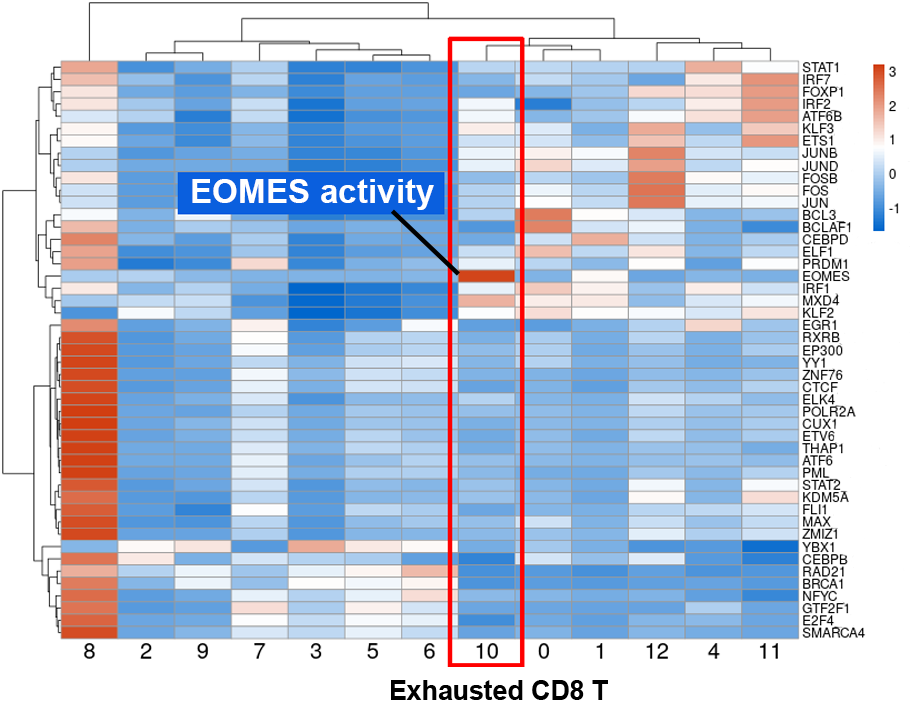

転写因子と共発現し転写因子の結合モチーフを有する遺伝子を転写因子の標的遺伝子として抽出し、その標的遺伝子の発現量から各T細胞サブセット(各クラスタ)で活性化している転写因子の推定を行いました。推定された転写因子活性をヒートマップで表示し、転写活性が高い場合を赤色、低い場合を青色で示しました。縦軸は転写因子、横軸は各T細胞サブセット(クラスタ番号)を示します。疲弊したT細胞で活性化する転写因子として知られるEOMESの転写活性が、疲弊したCD8 T細胞(クラスタ10、赤枠内)で高くなっています。
推定された転写因子の活性と発現分布
推定された転写因子EOMESの活性分布
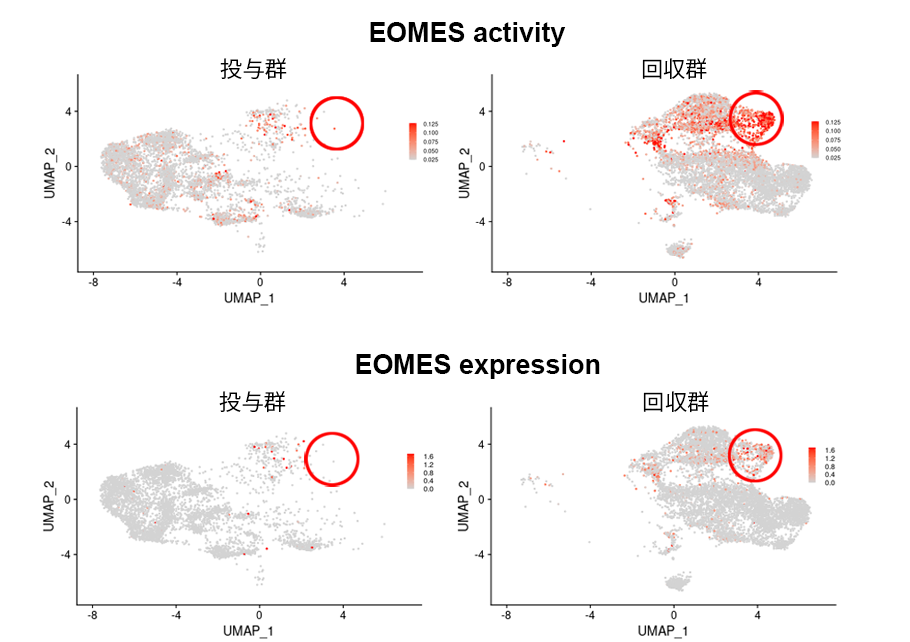

推定されたEOMESの転写活性(上段)と発現量(下段)を、群ごとに二次元に表示しました。転写活性及び発現量が高い細胞を赤色で示します。回収群では疲弊したCD8 T細胞(赤丸内)が多く、かつEOMESの転写活性が高いと推定されました。転写活性と発現量が対応しない領域が見られることから、発現量の制御以外に活性調節を受けていることが示唆されます。遺伝子の発現情報に加えて、当解析例で実施した転写因子活性を推定する解析や、シングルセルATAC-seqのようにエピジェネティックな状態を推定する解析を行うことで、生体内の現象をより詳細かつ高い信頼性で理解することが可能になると期待されます。
解析例11 心臓組織の細胞老化による細胞間相互作用の変化の検出
老化マウスの心臓で、老化した細胞が示す特徴的な発現プロファイル(Senescence-Associated Secretory Phenotype:SASP)を示す細胞が発信する細胞間相互作用の解析を実施し、若年マウスとの比較を行いました。
若年および老齢マウス心臓由来細胞のクラスタリング結果
若年および老齢マウス心臓由来細胞のクラスタリング結果
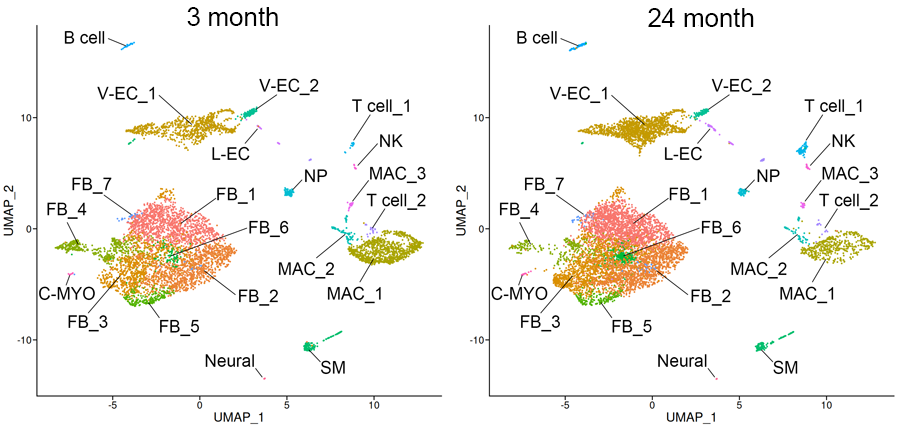

若年マウス(3 month, n=2)および老齢マウス(24 month, n=2)の心臓に由来する細胞(主に非筋細胞)をクラスタリングし、二次元上にプロットしました。老化による若干の細胞構成比の変化が見られましたが、老化に特徴的な細胞集団の消失や出現は見られませんでした。FB: fibroblast, V-EC: vascular endothelial cell, L-EC: lymphatic endothelial cell, SM: smooth muscle cell, MAC: macrophage, NP: neutrophil, NK: natural killer cell, C-MYO: cardiac myocyte.
老化によるSASP遺伝子群の発現変化 (Signature scoring)
老化によるSASP遺伝子群の発現変化 (Signature scoring)
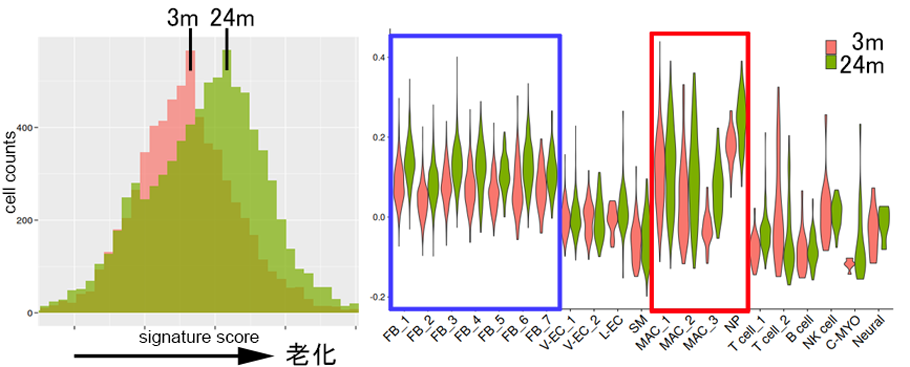

老化に特徴的な遺伝子群(SASP genes)の発現をスコアリングし、若年マウス(3m)と老齢マウス(24m)の全細胞を対象に細胞ごとのスコアをヒストグラムで比較しました。老齢マウスではヒストグラムが右に移動し、スコアが高い細胞が増加していました。また、各クラスタについて群別のスコアをviolin plotで表示すると、老齢マウスで線維芽細胞(青枠)、マクロファージおよび好中球(赤枠)などが高いスコアを示し、老化によるスコアの増加が見られました。
老化により細胞間相互作用に変化が見られた細胞タイプ
老化により細胞間相互作用に変化が見られた細胞タイプ
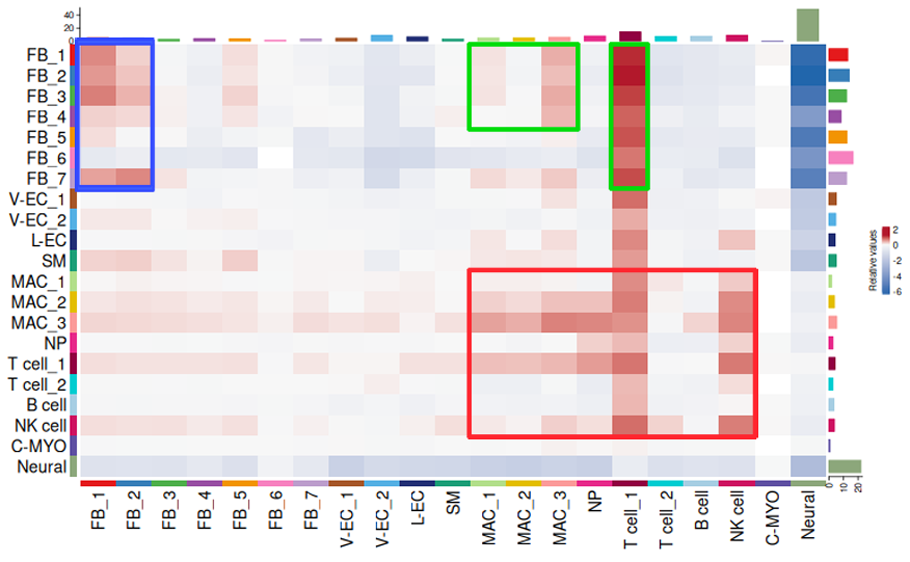

老化による細胞間相互作用の変化をヒートマップで表示しました。縦軸はシグナルの発信細胞、横軸は標的細胞を表しており、相互作用が増加した組合せを赤色、減少した組み合わせを青色で表示しました。線維芽細胞が発信および標的細胞となっている相互作用(青枠)、線維芽細胞が発信細胞、免疫系細胞が標的細胞となっている相互作用(緑枠)、免疫系細胞が発信および標的細胞となっている細胞間相互作用(赤枠)が大きく変化していました。
老化による線維芽細胞およびマクロファージが発信する細胞間相互作用の変化
老化による線維芽細胞およびマクロファージが発信する細胞間相互作用の変化
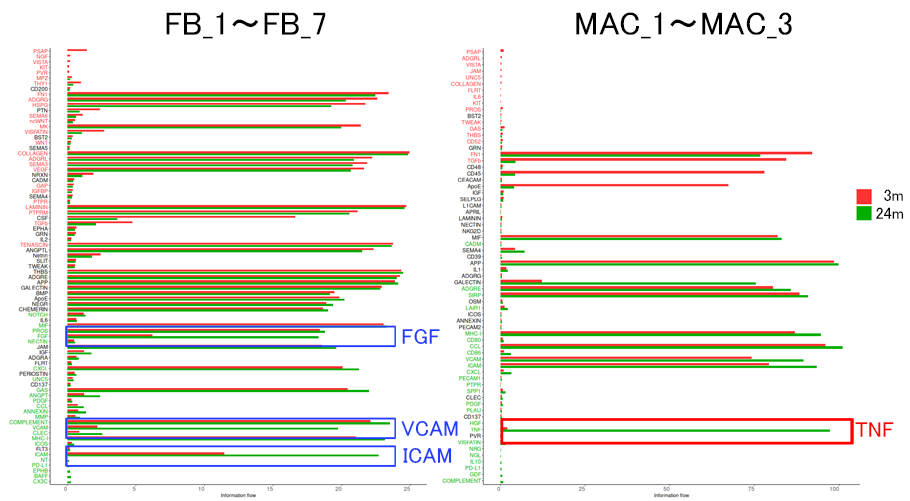

老化による細胞間相互作用パスウェイの変化を解析しました。縦軸に細胞間相互作用パスウェイ、横軸に各パスウェイの相互作用量をとり、若年マウスを赤色、老齢マウスを緑色の棒グラフで示しました。線維芽細胞(FB_1, 2, 3, 4, 5, 6, 7)を発信細胞とするパスウェイではFGF、VCAM、ICAM(左図、青枠)が、マクロファージ(MAC_1, 2, 3)を発信細胞とするパスウェイではSASPの代表的因子であるTNF(右図、赤枠)の相互作用量が老化により増加していました。
老化によりTNFシグナルが増加した細胞
老化によりTNFシグナルが増加した細胞
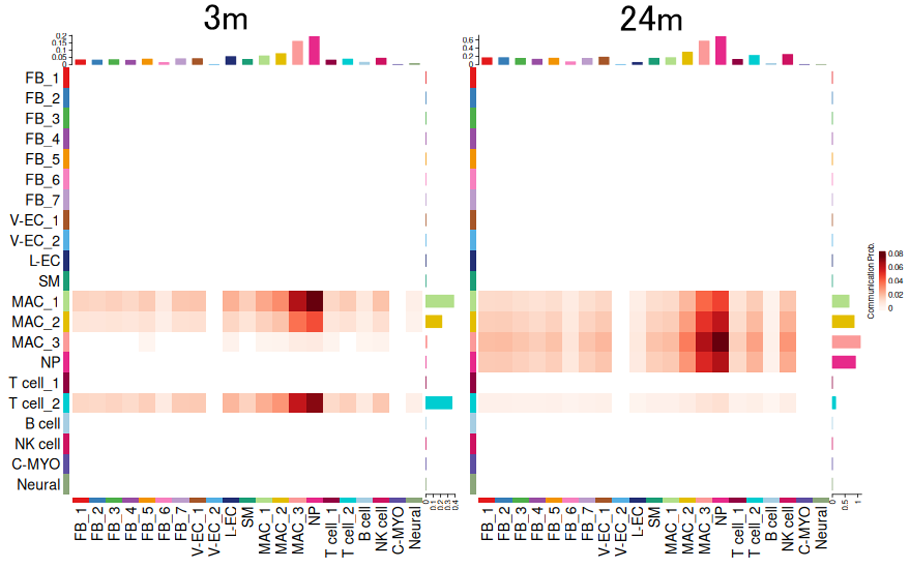

TNFシグナルの相互作用の強さをヒートマップで示しました。縦軸は発信細胞、横軸は標的細胞を表します。マクロファージ(MAC_2, 3)および好中球(NP)を発信細胞とするTNFシグナルが老化により増加していました(右図)。標的細胞側には顕著な変化は見られませんでした。老化により、一部のマクロファージおよび好中球の性質が変化し、TNFシグナルが増加した可能性が考えられました。
老化によるTNFおよびTNF受容体の発現レベルの変化
老化によるTNFおよびTNF受容体の発現レベルの変化
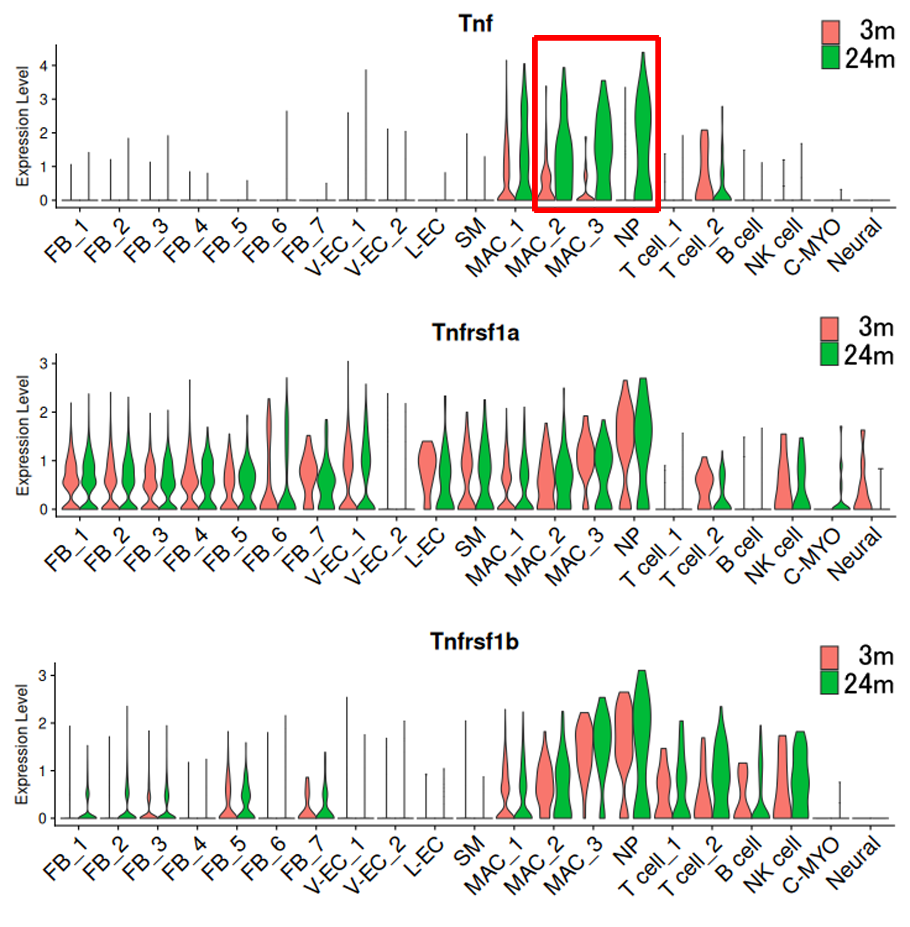

各クラスタのTNFおよびTNF受容体(Tnfrsf1a, Tnfrsf1b)発現量を群別にバイオリンプロットで表示しました。老化マウスのマクロファージ(MAC_2, MAC_3)および好中球(NP)でTNFの発現量が顕著に増加していました(上段、赤枠)。TNF受容体は老化による発現量の大きな変化は見られませんでした(中段、下段)。老化によるTNFシグナルの増加は、主にリガンドであるTNFの発現量増加に由来すると考えられました。
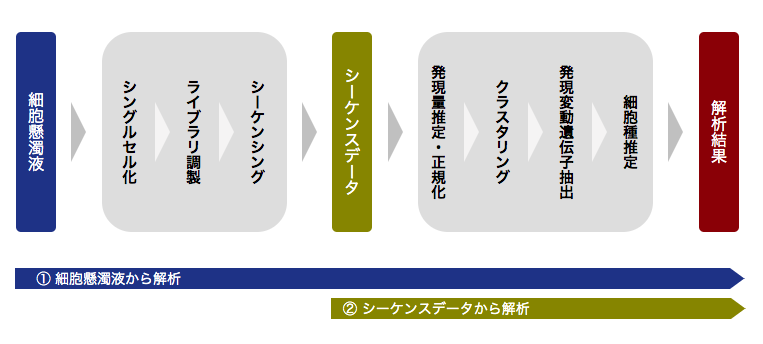
解析フロー
細胞懸濁液
シングルセル化
ライブラリ調製
シーケンシング
シーケンスデータ
発現量推定・正規化
クラスタリング
細胞タイプ推定
高次解析
解析結果
受入物
| ① 細胞懸濁液から解析 | ② シーケンスデータから解析 | |
|---|---|---|
| 受入物 | 細胞懸濁液 | シーケンスデータ(FASTQファイル) |
| 受入条件 |
【 生細胞の場合 】
【 凍結細胞の場合 】
※死滅しやすい細胞や臨床検体はこちら
|
※上記のほか様々なシングルセル化、ライブラリ調製法で取得したシーケンスデータに対応
|
| その他 |
【 全般 】
【 生細胞の場合 】
【 凍結細胞の場合 】
|
以下の情報をお知らせください
|

解析条件
- 解析対象細胞数(目安): 数百細胞~100万細胞
- 細胞当たりのリード数 : 研究目的に最適な条件をご提案します。
- 解析システム : ご要望に合わせてご提案します。
- データ解析 : 研究目的に最適な手法で実施します(Batch effect等の各種補正対応)
納品物
① 細胞懸濁液から解析- 解析報告書
- シーケンスデータ
- 細胞クラスタリング画像データ
- 各クラスタの細胞数データ
- 各種遺伝子の発現データ(発現量、発現分布)
- 各種遺伝子リストなど
- 解析報告書
- 細胞クラスタリング画像データ
- 各クラスタの細胞数データ
- 各種遺伝子の発現データ(発現量、発現分布)
- 各種遺伝子リストなど
※実際の納品物はご依頼内容により変わります。
価格・納期
ご依頼の解析内容により異なりますので、詳細はこちらよりお問い合わせください。
ご注意事項
受入物を品質検査した結果、解析をお引き受けできない場合があります。
お問い合わせ
こちらよりお問い合わせください。
専門技術者が原則24時間以内にご連絡します。(土日祝日は除く)
参考文献
Treutlein et al. “Dissecting direct reprogramming from fibroblast to neuron using single-cell RNA-seq” Nature vol.534, 391–395, 2016. doi: 10.1038/nature18323.
Zheng, C. et al. “Landscape of Infiltrating T Cells in Liver Cancer Revealed by Single-Cell Sequencing.” Cell 169, 1342-1356.e16 (2017). doi: 10.1016/j.cell.2017.05.035.
Semrau, S. et al. “Dynamics of lineage commitment revealed by single-cell transcriptomics of differentiating embryonic stem cells.” Nature Communications 8, (2017). doi: 10.1038/s41467-017-01076-4.
Krutzik, S. R. et al. “TLR activation triggers the rapid differentiation of monocytes into macrophages and dendritic cells.” Nature Medicine 11, 653–660 (2005). doi: 10.1038/nm1246.
Tang-Huau, T.-L. et al. “Human in vivo-generated monocyte-derived dendritic cells and macrophages cross-present antigens through a vacuolar pathway.” Nature Communications 9, (2018). doi: 10.1038/s41467-018-04985-0.
Sharir, A. et al. “A large pool of actively cycling progenitors orchestrates self-renewal and injury repair of an ectodermal appendage.” Nature Cell Biology 21, 1102–1112 (2019). doi: 10.1038/s41556-019-0378-2.
Schafflick, D. et al. Integrated single cell analysis of blood and cerebrospinal fluid leukocytes in multiple sclerosis. Nature Communications 11, (2020). DOI: 10.1038/s41467-019-14118-w
Hrvatin, S. et al. “Single-cell analysis of experience-dependent transcriptomic states in the mouse visual cortex.” Nature Neuroscience 21, 120–129. (2018). doi: 10.1038/s41593-017-0029-5
Guo, L. et al. (2019). Resolving Cell Fate Decisions during Somatic Cell Reprogramming by Single-Cell RNA-Seq. Molecular Cell 73, 815-829.e7. 10.1016/j.molcel.2019.01.042.
Huang, Y. et al. “Inhibition of CD38 enzymatic activity enhances CAR-T cell immune-therapeutic efficacy by repressing glycolytic metabolism.” Cell Reports Medicine 5, 101400 (2024). doi.org/10.1016/j.xcrm.2024.101400
*1 別途ご相談ください。データにより解析できない場合があります。
*2 ChromiumシステムTMは、10X Genomics社の登録商標です。
*3 RhapsodyシステムTMは、BD社の登録商標です。
*4 Uniform Manifold Approximation and Projection
*5 t-distributed Stochastic Neighbor Embedding
*6 ICELL8システムは、Takara Bio社の登録商標です。
*7 C1システムは、Fluidigm社の登録商標です。
