シングルセルレパトア解析+scRNA-seq解析
シングルセルレパトア解析+シングルセルRNA-seq解析とは
T細胞/B細胞の抗原受容体(TCR/BCR)を構成する遺伝子ペアを同定するレパトア解析と、網羅的な遺伝子の発現定量を行うRNAシーケンス解析を1細胞単位で同時に行うことにより、細胞のタイプや状態とTCR/BCRの遺伝子ペアの情報を紐付けた解析が可能です。
獲得免疫系は、T細胞やB細胞が中心となって多様な抗原に対して特異的に応答します。T cell receptor (TCR)およびB cell receptor (BCR)と呼ばれる抗原受容体は、遺伝子再構成などにより、TCRでは1018、BCRでは1014を超える膨大な多様性を確保しています。抗原受容体のバリエーションやそれによって特徴付けられる免疫細胞集団の多様性を免疫レパトアと呼びます。レパトア解析は、白血病や骨髄移植の評価、がんや自己免疫疾患における免疫応答の解析などに有用です。
1つの細胞が発現する抗原受容体は原則として1種類であることから、抗原受容体が同一の細胞はクローン(clone)であると考えられ、各サブユニット遺伝子の構造はクロノタイプ(clonotype)と呼ばれています。例えばT細胞では、TCRのサブユニットであるα鎖(TRA)、β鎖(TRB)の遺伝子をペアで決定すると、その細胞が発現しているTCRが決定されます。シングルセルレパトア解析では、1細胞ごとに配列情報を取得するため、各クロノタイプを持つ細胞を定量的に評価できるだけでなく、バルク(集団)のレパトア解析では困難な、各サブユニットのペアを決定した状態で免疫レパトアを網羅的に解析できます。RNA-seq解析と組み合わせることにより、特定のタイプや状態の細胞の間でのレパトアを比較することが可能です。
※受託解析サービスの詳細はこちら
用途
- 1細胞レベルでのクロノタイプ同定と遺伝子発現解析
- T細胞およびB細胞集団の異質性(heterogeneity)評価
- 特定の細胞タイプでのクロノタイプ分布の解析
- 特定のクロノタイプでの遺伝子発現解析
- 多数の細胞中に混在するT細胞およびB細胞のクロノタイプ同定
- 細胞タイプ推定*1(未知の細胞タイプ検出を含む)
※関連する解析サービス
シングルセルRNA-seq解析 >
ホルマリン固定後のシングルセルトランスクリプトーム解析 >
高感度なシングルセル完全長total RNA-seq解析 >
シングルセル空間トランスクリプトーム解析 >
スポット型空間トランスクリプトーム解析 >
このような方に
- 参考論文と同じような解析を自分のサンプルで実施したい方
- 自分の研究目的に最適な実験デザインの検討から始めたい方
- 簡易解析による解析結果と生物学的な仮説との間に相違があり、結果の解釈にお困りの方
- TCRのα鎖およびβ鎖の両方が検出された信頼性の高い細胞のデータのみで解析したい方
- 共同研究先の対応スピードや、一方向的な情報科学的解析手法にお悩みの方
- 最適なデータ解析手法や適切な統計手法の選択と実施に不安がある方
当社の特徴
- 共同研究タイプの受託解析サービス(定型的な受託解析ではありません)
- ウェット実験とデータ解析の両分野の経験豊富な専門技術者が直接対応
- TCRのα鎖およびβ鎖の両方が検出された細胞のデータのみを抽出して信頼性の高い解析を実施
- 生物学的意義を踏まえたデータ解析サービス(詳細はこちら)
- 解析結果の統計学的および生物学的解釈のサポート
※「当社が選ばれる5つの理由」はこちら
解析例1 腫瘍浸潤T細胞のクラスタリングとレパトア解析
淡明細胞型腎細胞がん患者の末梢血リンパ球と腫瘍浸潤リンパ球に対して、シングルセルレパトア解析と遺伝子発現解析を同時に行ったシーケンスデータを使用してマルチオミクス解析を実施しました。RNA発現プロファイルに基づいて細胞を分類し、T細胞のタイプとレパトアの情報を紐付けて解析を行いました。
腫瘍浸潤リンパ球と末梢血リンパ球のクラスタリング結果(23,489 cells)
腫瘍浸潤リンパ球と末梢血リンパ球のクラスタリング結果(23,489 cells)
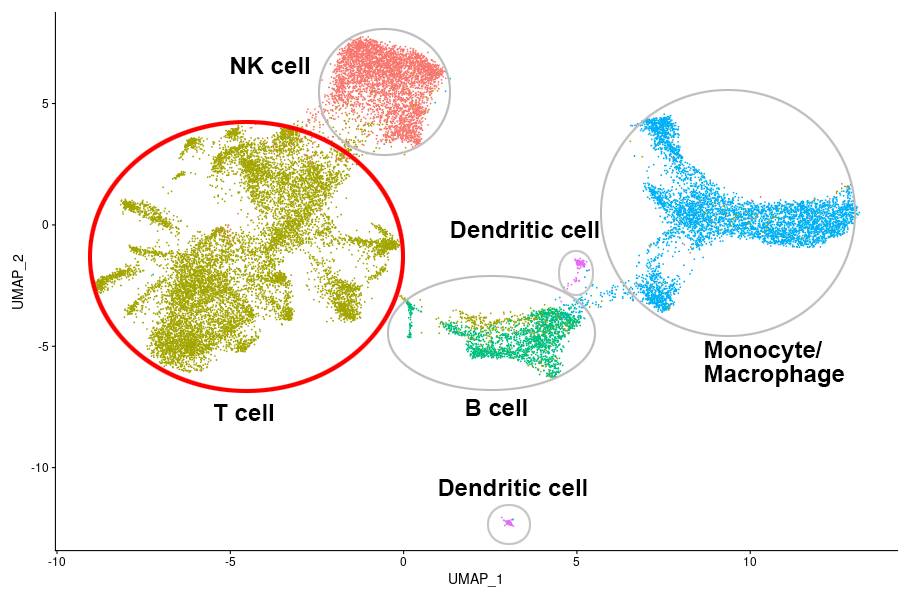

RNA発現プロファイルに基づいて細胞をクラスタリングし、細胞タイプを推定した結果を二次元表示しました。赤丸で囲ったクラスタがT細胞と推定されました。T細胞のうち、TCRのα鎖(TRA)およびβ鎖(TRB)の両方が検出された細胞のデータをシングルセルレパトア解析に使用しました。
クロノタイプごとのT細胞クローン数
クロノタイプごとのT細胞クローン数
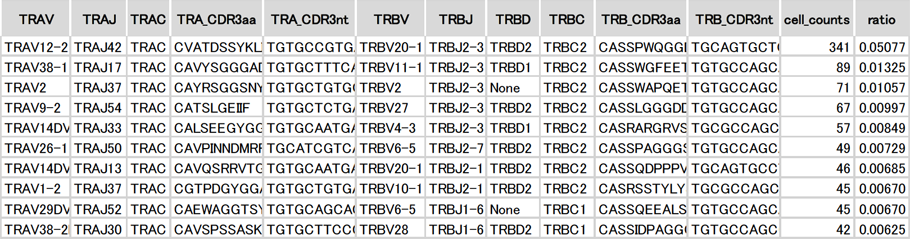

T細胞の各クローンを細胞数順にリスト化し、TCR遺伝子とCDR3領域*2の塩基配列の詳細を表示しました。バルク(集団)のレパトア解析では、TCR遺伝子配列の出現割合を細胞数の割合とみなすため増幅バイアスが問題となりますが、シングルセルレパトア解析では、細胞ごとに配列を決定し、細胞数自体をカウントするため増幅バイアスの影響を受けません。
V-J遺伝子構成割合グラフ
V-J遺伝子構成割合グラフ
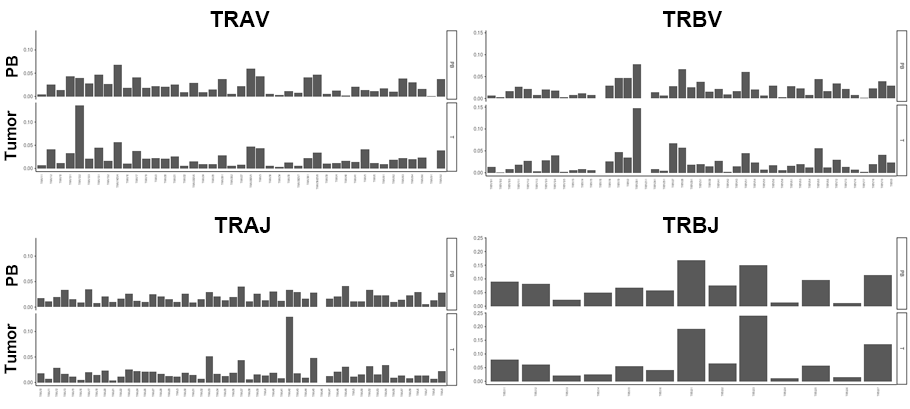

TCR遺伝子を構成するVおよびJ各遺伝子の構成割合を末梢血T細胞(PB)と腫瘍浸潤T細胞(Tumor)で比較すると、全体的な割合の類似が認められました。腫瘍浸潤T細胞において、一部の遺伝子の割合が増えているのは、腫瘍組織中でT細胞の増殖が起こったためと推定されます。グラフの縦軸はV-J各遺伝子の構成割合、横軸はV-J各遺伝子を示します。
V-J遺伝子の構成割合(Heatmap)
V-J遺伝子の構成割合(Heatmap)
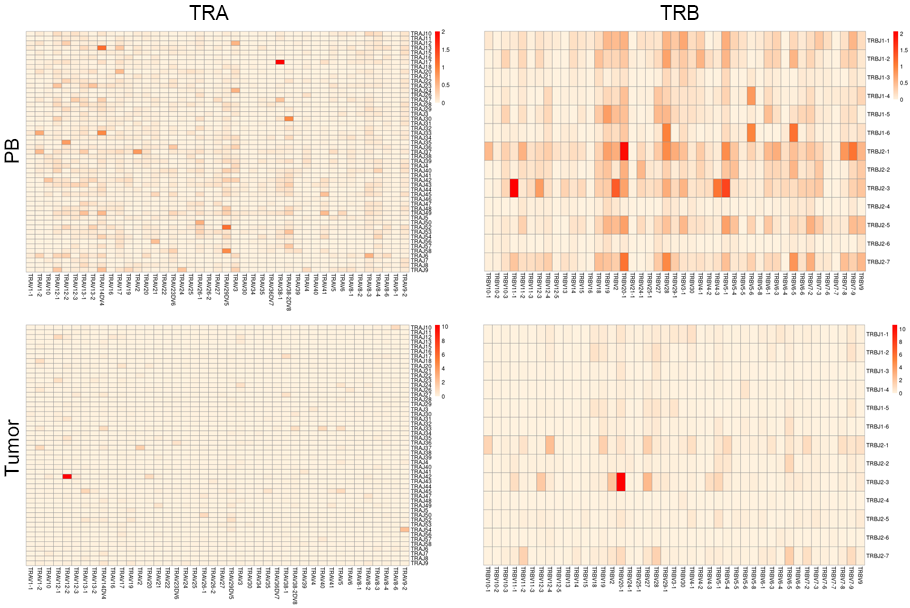

V-J遺伝子の構成割合をヒートマップで表示しました。腫瘍浸潤T細胞(下段)では、腫瘍組織中で増殖したと考えられるT細胞が見られました。ヒートマップの縦軸はJ遺伝子、横軸はV遺伝子を示します。
CDR3のアミノ酸配列(TRA+TRB)ごとのT細胞クローン数
CDR3のアミノ酸配列(TRA+TRB)ごとのT細胞クローン数
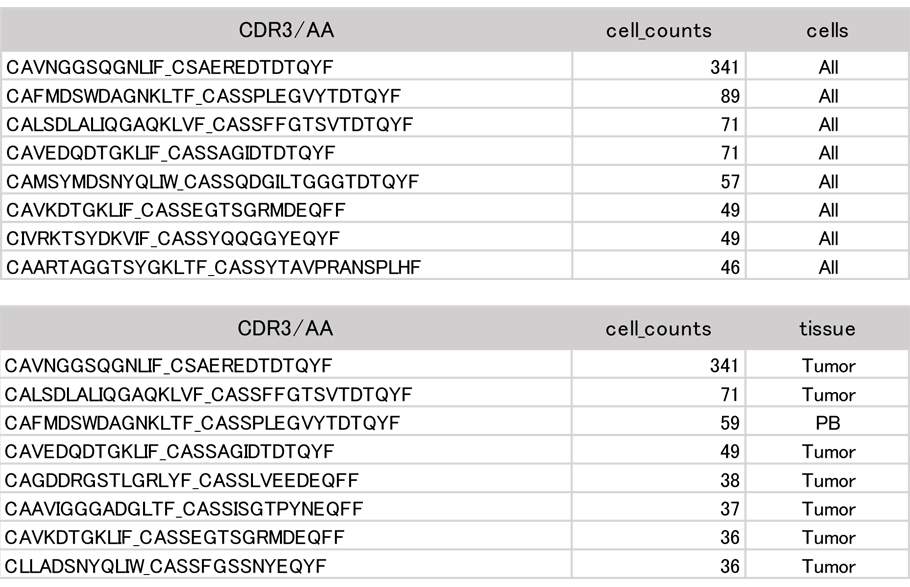

CDR3は、TCRの抗原認識部位を構成する領域の中でも特異性に対する寄与が大きいとされています。CDR3のアミノ酸配列を細胞数の多い順にリスト表示しました。上段は全T細胞、下段は由来組織毎に細胞数をまとめたリストです。
CDR3クロノタイプの細胞数上位10が占める割合
CDR3クロノタイプの細胞数上位10が占める割合
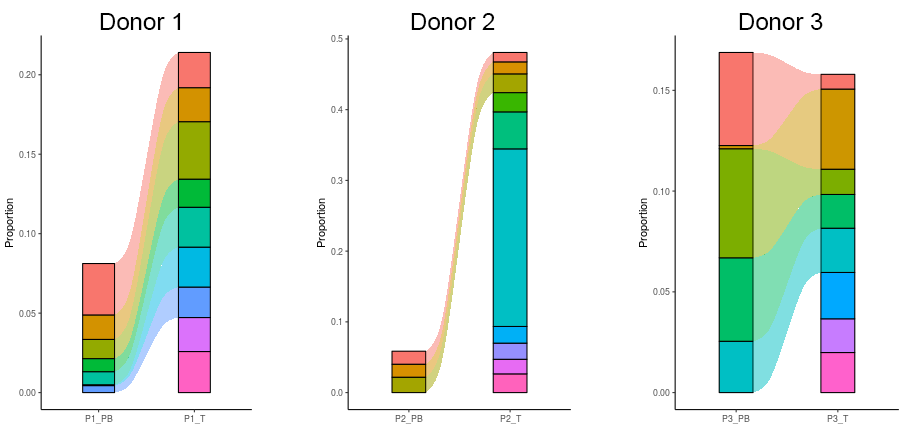

各CDR3クロノタイプを持つT細胞クローンについて、細胞数上位10の割合を患者ごとにグラフ化しました。各グラフ左側に末梢血T細胞、右側に腫瘍浸潤T細胞を示しました。末梢血T細胞で見られたクロノタイプが腫瘍浸潤T細胞でも見られたことから、末梢血中で増加していたT細胞が腫瘍に移行したと推定されました。また、進行したステージの患者(中央、Donor 2)では腫瘍浸潤T細胞のみで見られるクロノタイプが多く検出され、腫瘍組織中で独立してT細胞の増殖が起きたと推定されました。
T細胞の再クラスタリング結果(12,269 cells)
T細胞の再クラスタリング結果(12,269 cells)
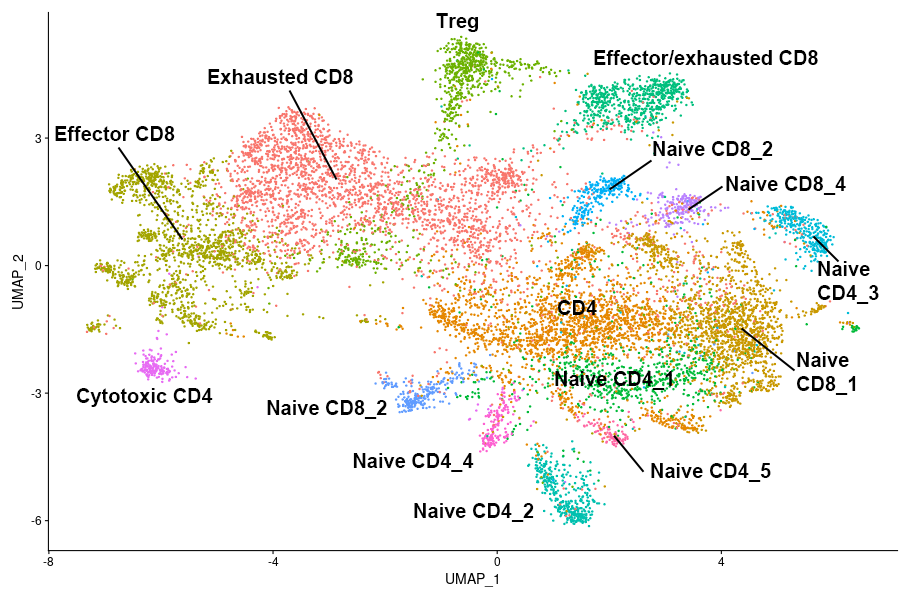

全細胞のクラスタリング結果から、T細胞のみを再度クラスタリングし、より詳細な各細胞タイプを推定しました。一部には疲弊した(exhausted)T細胞の集団が含まれていました。このようにシングルセルRNA-seq解析の結果からは、細胞のタイプや状態に関する情報を取得できます。
T細胞における各細胞タイプのクローン数の割合
T細胞における各細胞タイプのクローン数の割合
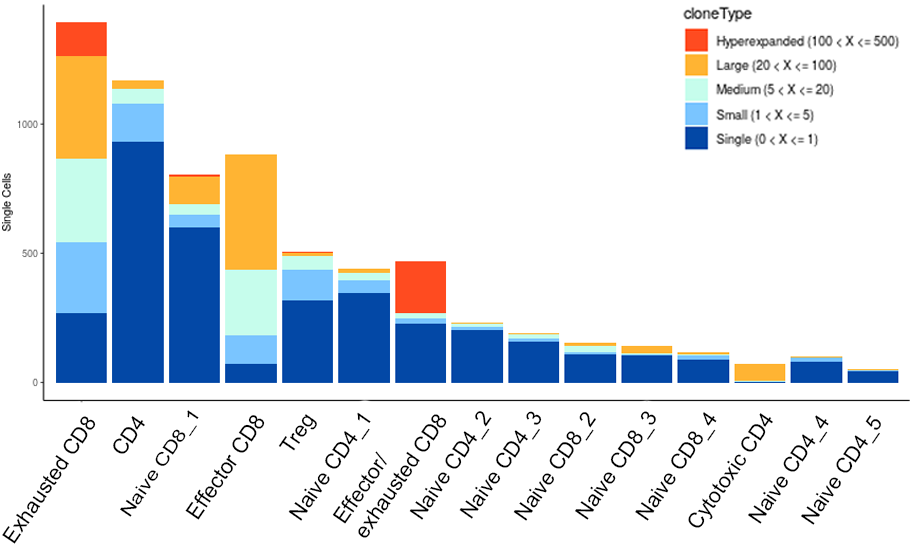

細胞タイプごとに、各T細胞クローンの細胞数の割合を表示しました。増殖したクローン(101細胞以上)を赤で示しました。特定の細胞タイプ(exhausted CD8, effector CD8, effector/exhausted CD8)で強く増殖が起こっていることが示されました。縦軸はTRA, TRB chainの両方が検出された細胞数、横軸は細胞タイプを示します。
T細胞における各細胞タイプのクローン数の分布
T細胞における各細胞タイプのクローン数の分布
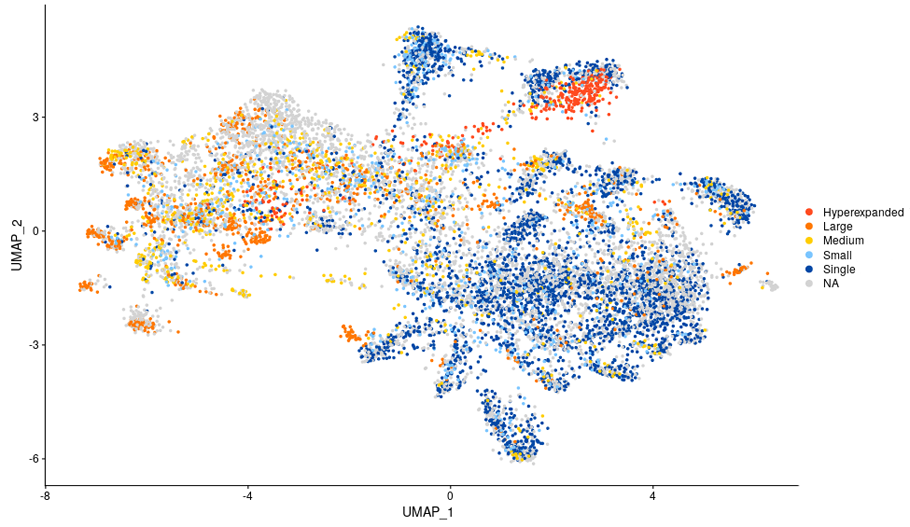

T細胞クローンを細胞数で色分けして分布を可視化しました。細胞数の多いクローンは赤で表示しました。exhausted CD8、effector/exhausted CD8で顕著な増殖が見られました。
exhausted CD8に特徴的な遺伝子の発現分布
exhausted CD8に特徴的な遺伝子の発現分布
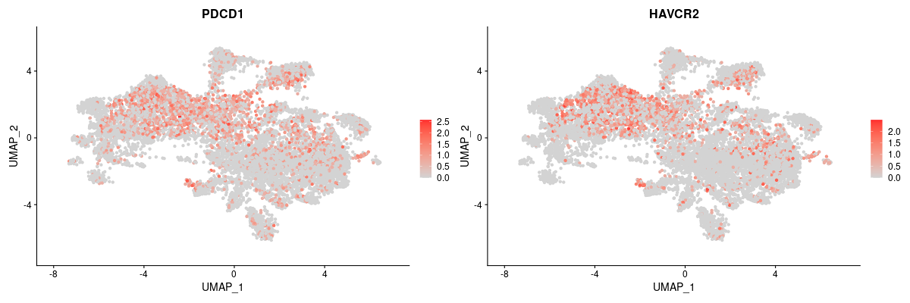

exhausted CD8で特徴的に発現するとされているPDCD1, HAVCR2の発現分布を示しました。このようにレパトア解析の結果と遺伝子発現情報を関連づけた解析が可能です。
末梢血T細胞と腫瘍浸潤T細胞クローン数の分布比較
末梢血T細胞と腫瘍浸潤T細胞クローン数の分布比較
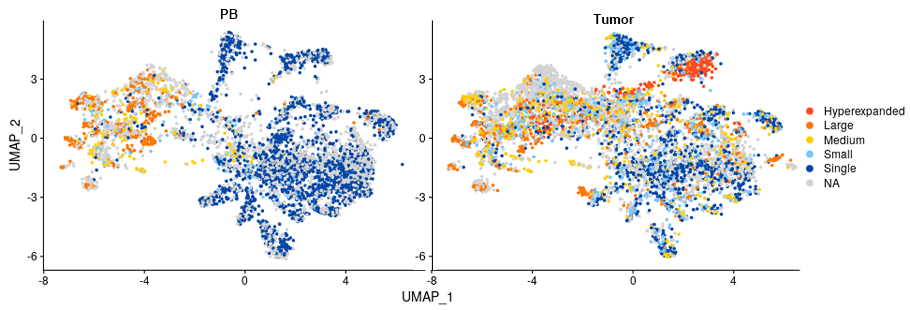

末梢血T細胞(左側)および腫瘍浸潤T細胞(右側)のクローン数の分布を表示しました。腫瘍浸潤T細胞では、赤で示したように細胞数の多いクローンが分布していました。
細胞数上位3のT細胞クローンの分布
細胞数上位3のT細胞クローンの分布
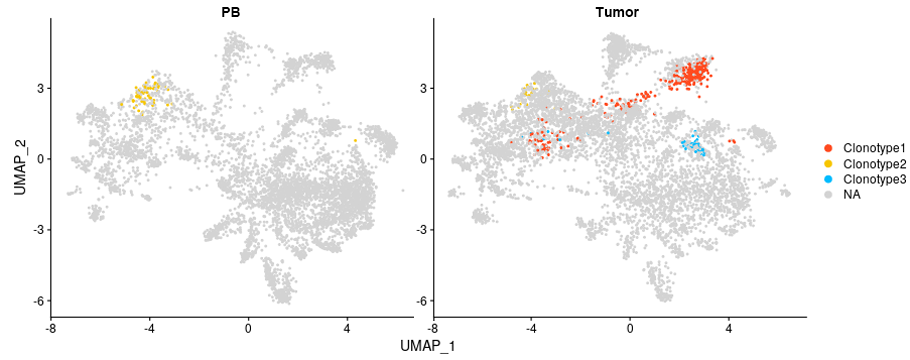

末梢血T細胞(左側)および腫瘍浸潤T細胞(右側)ごとに、細胞数上位3のT細胞クローンの分布を表示しました。細胞毎にクロノタイプの情報が関連付けられているため、特定のクロノタイプを指定して分布を表示することが可能です。
このように1細胞ごとにRNA発現プロファイルとレパトアの情報を同時に取得することにより、細胞のタイプや状態に関する情報と、レパトアの情報を紐付けて解析することが可能となりました。これにより、レパトアが変化した細胞集団を特定し、細胞のタイプや生理的な状態を推定することが可能になります。
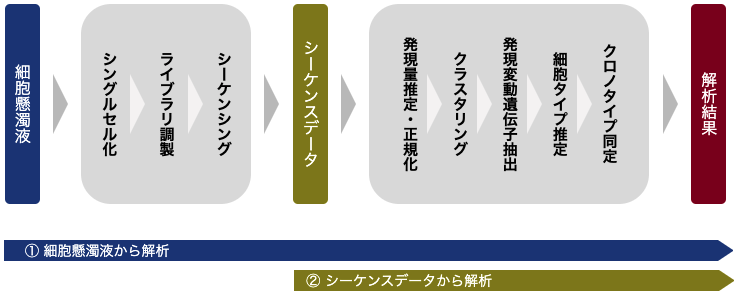
解析フロー
細胞懸濁液
シングルセル化
ライブラリ調製
シーケンシング
シーケンスデータ
発現量推定・正規化
クラスタリング
発現変動遺伝子抽出
細胞タイプ推定
クロノタイプ同定
解析結果
受入物
| ① 細胞懸濁液から解析 | ② シーケンスデータから解析 | |
|---|---|---|
| 受入物 | 細胞懸濁液 | シーケンスデータ(FASTQファイル) |
| 生物種 | ヒトまたはマウス | ヒトまたはマウス |
| 受入条件 |
【生細胞の場合】
【凍結細胞の場合】
|
|
| その他 |
【全般】
【生細胞の場合】
【凍結細胞の場合】
|
以下の情報をお知らせください
|

解析条件
- 解析対象細胞数(目安): 数百細胞~100万細胞
- 細胞当たりのリード数 : 研究目的に最適な条件をご提案します。
- データ解析 : 研究目的に最適な手法で実施します(Batch effect等の各種補正対応)
納品物
①細胞懸濁液から解析- 解析報告書
- シーケンスデータ
- 細胞クラスタリング画像データ
- 各クラスタの細胞数データ
- 各種遺伝子の発現データ(発現量、発現分布)
- 各種遺伝子リストなど
- TCR/BCR遺伝子構成リスト
- クロノタイプランクリスト
- CDR3アミノ酸配列ランクリスト
- V(D)J遺伝子構成比データ
- クローン数分布グラフ
- クローン数分布画像データ
- 解析報告書
- 細胞クラスタリング画像データ
- 各クラスタの細胞数データ
- 各種遺伝子の発現データ(発現量、発現分布)
- 各種遺伝子リストなど
- TCR/BCR遺伝子構成リスト
- クロノタイプランクリスト
- CDR3アミノ酸配列ランクリスト
- V(D)J遺伝子構成比データ
- クローン数分布グラフ
- クローン数分布画像データ
※実際の納品物はご依頼内容により変わります。
価格・納期
ご依頼の解析内容により異なりますので、詳細はこちらよりお問い合わせください。
ご注意事項
受入物を品質検査した結果、解析をお引き受けできない場合があります。
お問い合わせ
こちらよりお問い合わせください。
専門技術者が原則24時間以内にご連絡します。(土日祝日は除く)
参考文献
Borcherding, N., Vishwakarma, A., Voigt, A.P., Bellizzi, A., Kaplan, J., Nepple, K., Salem, A.K., Jenkins, R.W., Zakharia, Y., and Zhang, W. (2021). Mapping the immune environment in clear cell renal carcinoma by single-cell genomics. Commun Biol 4, 122.
https://doi.org/10.1038/s42003-020-01625-6
*2 CDR3 : complementarity determining region 3
