ChIP-seq受託解析
ChIP-seq解析とは
修飾ヒストンや転写調節因子に対する抗体を用いて、断片化された染色体DNAを含む複合体を免疫沈降し、目的タンパク質が結合しているクロマチンDNAを濃縮して塩基配列を決定します。クロマチン上の転写調節因子結合部位、ヒストン修飾部位を網羅的かつ高分解能で同定出来るため、結合モチーフ配列や転写複合体構造の推定に役立ちます。また、転写調節因子の結合部位近傍にある遺伝子を抽出し、目的の転写調節因子によって転写調節を受ける遺伝子群を網羅的に探索する事も可能です。
用途
- 転写調節因子の結合部位、結合モチーフ配列の推定
- ヒストン修飾部位の推定
- 目的の転写調節因子によって転写調節を受ける遺伝子群の網羅的探索
解析例
β-カテニンは、発生や形態形成に関わるWntシグナル経路の下流で転写調節因子として機能しています。Wnt3aで刺激したヒトES細胞に対して、β-カテニン抗体でChIP-seqを行ったデータを用いてマッピング、ピーク検出、結合モチーフ配列推定を行いました。
ピーク検出結果
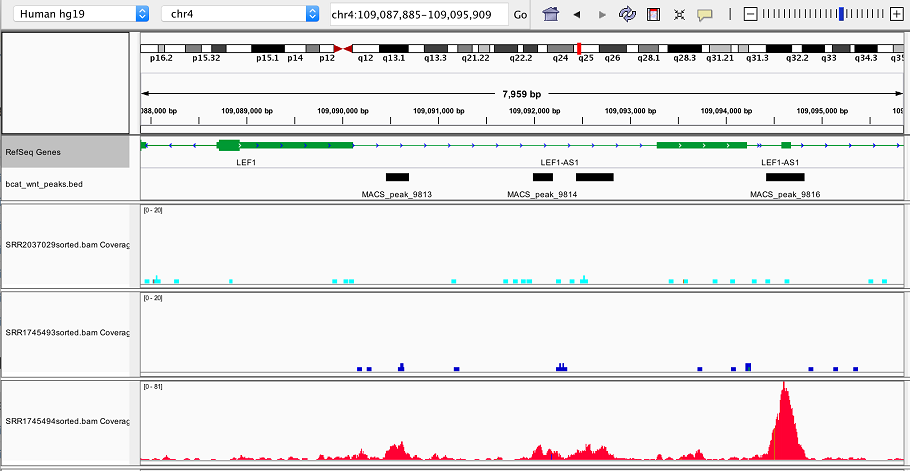
β-カテニン抗体によるChIP-seqデータを用いて、ピーク検出(ピークコール)を行った結果を図示しました。免疫沈降前(上段、水色)、Wnt3a刺激なし(中段、青)ではほとんどピークが見られないのに対して、Wnt3a刺激した細胞(下段、赤)では、β-カテニンによって転写が活性化されるLEF1遺伝子の上流にピークが検出されました*1 。
転写開始点に対する転写調節因子結合部位の可視化
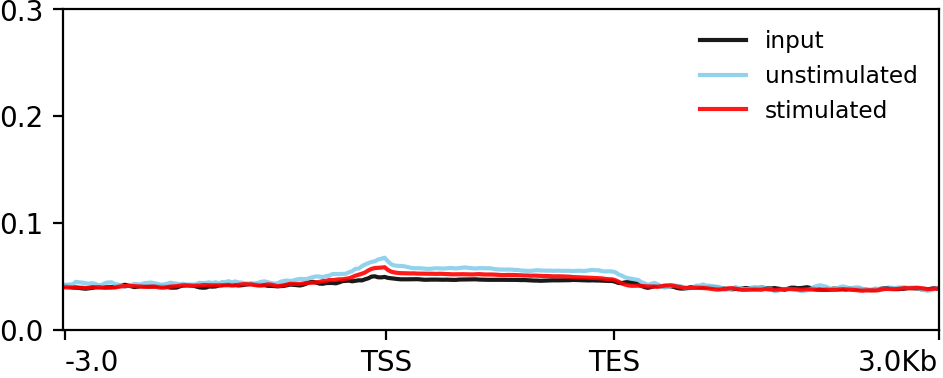
全遺伝子の転写開始点(TSS)に対してβ-カテニンの結合部位をプロットしました。β-カテニンの結合部位は特定の遺伝子の周辺に限られるため、全遺伝子を対象としたプロットではWnt3a刺激した細胞(stimulated)と未刺激細胞(unstimulated)との間に差は認められませんでした。input:免疫沈降前。
転写複合体因子同士の相対的な位置
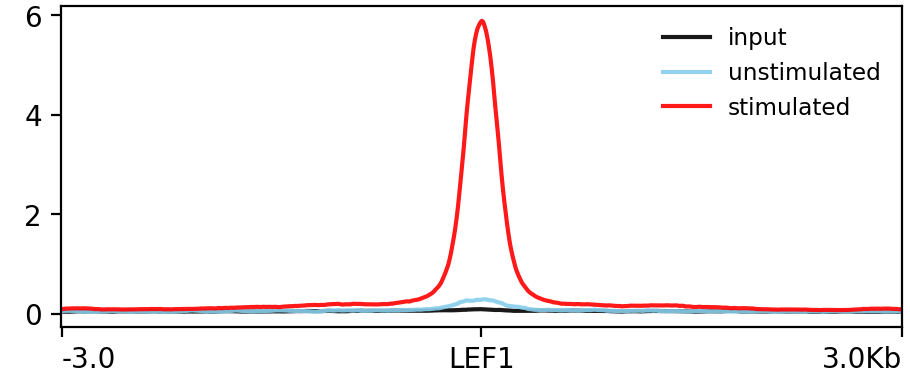
一方、β-カテニンと結合して転写を活性化するLEF1のDNA結合部位(中央)に対して、β-カテニン結合部位をプロットすると、Wnt3a刺激した細胞(stimulated)でのみLEF1結合部位にβ-カテニンが特異的に結合していることが示されました。
転写複合体因子同士の相対的な位置(Heatmap)
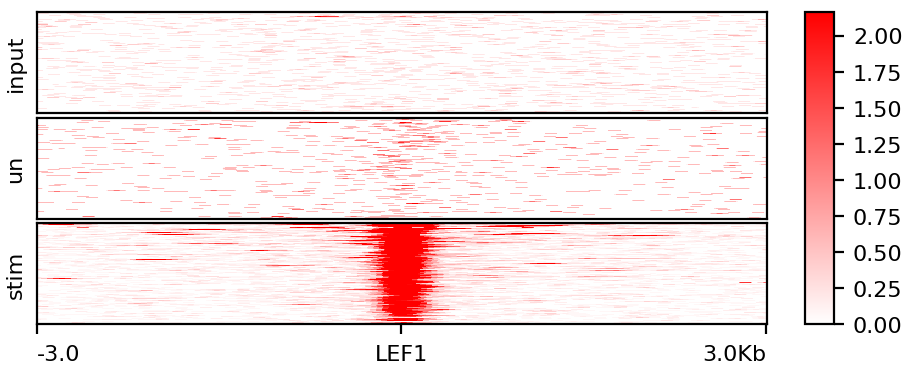
LEF1結合部位を中央にして、β-カテニンの結合部位をヒートマップで可視化しました。Wnt3a刺激した細胞(下段、stim)では、未刺激細胞(中段、un)に比べ、赤で示したβ-カテニンの結合がLEF1結合部位の周囲に集中しているのが確認できました。
結合モチーフ解析
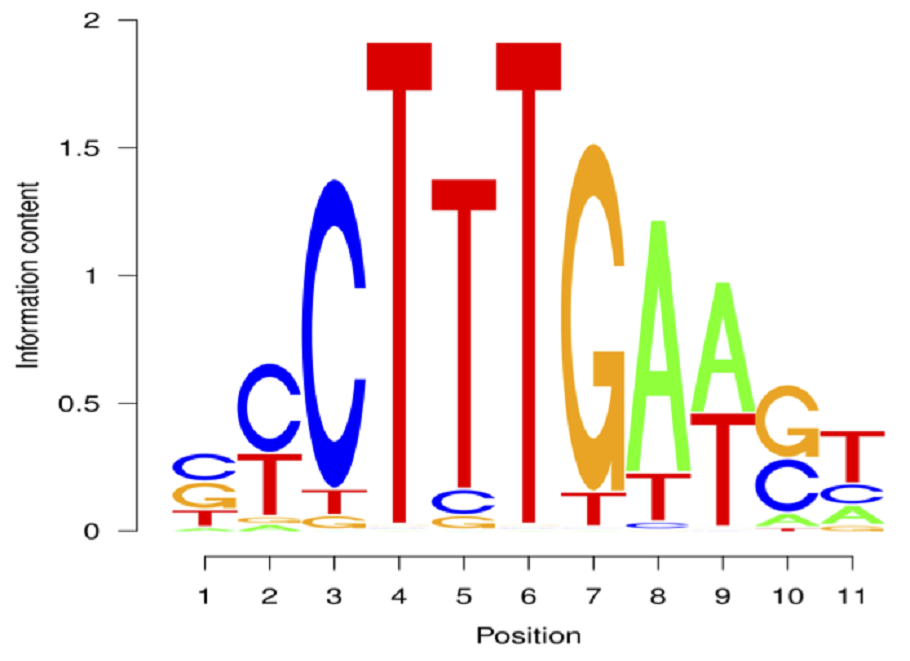
統計的に信頼性の高いピークに共通するモチーフ配列を探索した結果、β-カテニンと複合体を形成して転写を活性化するTCF7, LEF1に類似した結合モチーフが見出されました。転写複合体として同じ部位に結合していることを反映した結果であると推定されます。出現頻度が高い塩基ほど大きく表示されます。
特徴
- 生物学的意義を踏まえたデータ解析
- 論文、学会発表に使用できる各種グラフの作成まで一貫してサポート
- スピーディーな解析結果報告(6営業日~、データ解析のみ時)
- 統計学の専門用語を極力排除した分かり易い説明
- 解析結果の統計学的および生物学的解釈のサポート
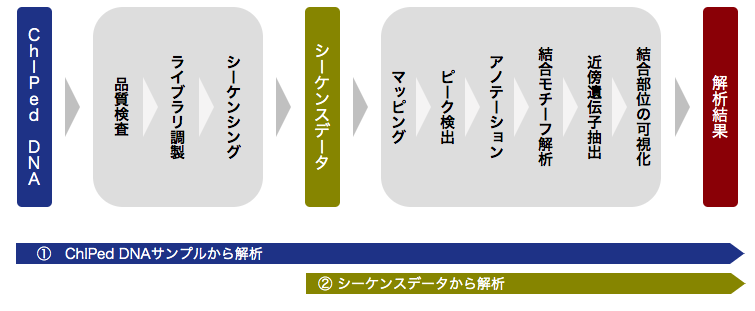
解析フロー
ChIPed DNA
品質検査
ライブラリ調製
シーケンシング
シーケンスデータ
マッピング
ピーク検出
アノテーション
結合モチーフ解析
近傍遺伝子抽出
結合部位の可視化
解析結果
受入物
| ① ChIPed DNAサンプルから解析 | ② シーケンスデータから解析 | ||
|---|---|---|---|
| 受入物 | ChIP回収した断片化DNA (断片サイズ:200〜600bp程度) |
シーケンスデータ (FASTQファイル) | |
| 受条件入 | 必要量 | 10ng以上(1 ng/ul以上、10ul以上) |
※上記のほか、様々なシーケンサーで取得したデータに対応
|
| 溶媒 | TE buffer等 | ||
| 品質 | A 260/280 >= 1.8 | ||
| ご注意事項 |
|
以下の情報をお知らせください
|
|

価格・納期
ご依頼の解析内容により異なりますので、詳細はこちらよりお問い合わせください。
サービスの流れ
こちらをご覧ください。
ご注文方法
こちらよりお問い合わせください。
ご注意事項
受入データを品質検査した結果、解析をお引き受けできない場合があります。
参考文献
Conchi Estarás et al. “YAP repression of the WNT3 gene controls hESC differentiation along the cardiac mesoderm lineage.” GENES & DEVELOPMENT 31:2250–2263, 2017. doi: 10.1101/gad.307512.117.
*1 この実験ではサンプル間の正規化が行われていないため、異なるサンプル間での定量性は保証されません。
